WikiDer > Микромассив олигонуклеотидов, специфичных для метилирования
Микромассив олигонуклеотидов, специфичных для метилирования, также известный как Микрочип MSO, был разработан как метод отображения эпигенетическое метилирование изменения в ДНК из рак клетки.[1]
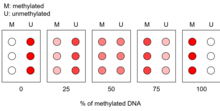
Общий процесс начинается с модификации ДНК с помощью бисульфит, специально для преобразования неметилированный цитозин в CpG сайты к урацилу, оставляя метилированный цитозины нетронутые.[1] Интересующий модифицированный участок ДНК амплифицируется с помощью ПЦР и во время этого процесса урацилы превращаются в тимин. В ампликоны помечены флуоресцентный краситель и гибридизирован с олигонуклеотидом зонды которые закреплены на предметном стекле.[2] Зонды по-разному связываются с остатками цитозина и тимина, что в конечном итоге позволяет различать метилированные и неметилированные сайты CpG соответственно.[1]
Строится калибровочная кривая и сравнивается с результатами на микрочипе амплифицированных образцов ДНК. Это позволяет в целом количественно оценить долю метилирования, присутствующего в интересующей области.[3]
Этот метод микроматрицы был разработан Тимом Хуэй-Мин Хуанг и его лабораторией и был официально опубликован в 2002 году.[1]
Значение для исследований рака
Эта статья нужно больше медицинские справки за проверка или слишком сильно полагается на основные источники. (Ноябрь 2019) |
Рак клетки часто развиваются атипично метилирование шаблоны, на CpG сайты в покровителях гены-супрессоры опухолей. Высокий уровень метилирования на промоторе приводит к подавлению регуляции соответствующих генов и характерен для канцерогенез. Это одно из наиболее устойчивых изменений, наблюдаемых в опухолевых клетках на ранних стадиях.[1] Микромассив олигонуклеотидов, специфичных для метилирования, позволяет с высоким разрешением и высокой пропускной способностью обнаруживать многочисленные события метилирования на промоторах нескольких генов. Следовательно, этот метод может использоваться для обнаружения аберрантного метилирования в промоторах супрессоров опухолей на ранней стадии и был использован при раке желудка, толстой кишки и многих других.[4][5] Поскольку он позволяет обнаруживать наличие атипичных метилирований в раковых клетках, его также можно использовать для выявления основной причины злокачественного новообразования, будь то мутации в хромосомах или эпигенетические модификации, а также уровни транскрипции генов-супрессоров опухолей. под действием.[2][6] Интересное использование этого микроматрицы включает в себя специфическую классификацию рака, основанную только на паттернах метилирования, например, различение классов лейкемия, предполагая, что разные классы рака демонстрируют относительно уникальные паттерны метилирования.[7] Этот метод также был предложен для мониторинга лечение рака, которое включает изменение паттернов метилирования в мутантных раковых клетках.[2]
Рекомендации
- ^ а б c d е Gitan RS, Shi H, Chen CM, Yan PS, Huang TH (январь 2002 г.). «Микроматрица специфических для метилирования олигонуклеотидов: новый потенциал для высокопроизводительного анализа метилирования». Геномные исследования. 12 (1): 158–64. Дои:10.1101 / гр.202801. ЧВК 155260. PMID 11779841.
- ^ а б c Ши Х, Майер С., Ниммрих И., Ян П.С., Колдуэлл CW, Олек А., Хуанг Т.Х. (январь 2003 г.). «Микроматрица на основе олигонуклеотидов для анализа метилирования ДНК: принципы и приложения». Журнал клеточной биохимии. 88 (1): 138–43. Дои:10.1002 / jcb.10313. PMID 12461783. S2CID 41907903.
- ^ Ян П.С., Вэй Ш., Хуан Т.Х. (2004). "Метилирование-специфический олигонуклеотидный микрочип". В Толлефсбол ТО (ред.). Протоколы эпигенетики. Методы молекулярной биологии. 287. Humana Press. С. 251–60. Дои:10.1385/1-59259-828-5:251. ISBN 9781592598281. PMID 15273417.
- ^ Хоу П, Шен Джи, Джи МДж, Хе Нью-Йорк, Лу Чж (декабрь 2004 г.). «Метод на основе микрочипов для обнаружения изменений метилирования 5'-CpG-островков гена p16 (Ink4a) в карциномах желудка». Всемирный журнал гастроэнтерологии. 10 (24): 3553–8. Дои:10.3748 / wjg.v10.i24.3553. ЧВК 4611991. PMID 15534905.
- ^ Mund C, Beier V, Bewerunge P, Dahms M, Lyko F, Hoheisel JD (апрель 2005 г.). «Матричный анализ паттернов метилирования геномной ДНК промотора гена супрессора опухолей p16INK4A в клеточных линиях карциномы толстой кишки». Исследования нуклеиновых кислот. 33 (8): e73. Дои:10.1093 / nar / gni072. ЧВК 1087791. PMID 15860770.
- ^ Ю. Ю., Паранджпе С., Нельсон Дж., Финкельштейн С., Рен Б., Коккинакис Д. и др. (Февраль 2005 г.). «Высокопроизводительный скрининг статуса метилирования генов при раке простаты с использованием массива метилирования олигонуклеотидов». Канцерогенез. 26 (2): 471–9. Дои:10.1093 / carcin / bgh310. PMID 15485992.
- ^ Адорьян П., Дистлер Дж., Липшер Э., Модель F, Мюллер Дж., Пелет С. и др. (Март 2002 г.). «Прогнозирование и обнаружение класса опухоли с помощью анализа метилирования ДНК на основе микрочипов». Исследования нуклеиновых кислот. 30 (5): 21e – 21. Дои:10.1093 / nar / 30.5.e21. ЧВК 101257. PMID 11861926.
внешняя ссылка
| Викискладе есть медиафайлы по теме Микромассив олигонуклеотидов, специфичных для метилирования. |
- Ресурсы, информация и специальные протоколы для анализа метилирования ДНК
- Программное обеспечение для анализа метилирования ДНК
| Этот генетика статья - это заглушка. Вы можете помочь Википедии расширяя это. |
| Этот онкология статья - это заглушка. Вы можете помочь Википедии расширяя это. |