WikiDer > Многовидовой процесс слияния
Многовидовой процесс слияния представляет собой модель стохастического процесса, которая описывает генеалогические отношения для выборки ДНК последовательности взяты от нескольких видов.[1] Он представляет собой применение коалесцентная теория в случае нескольких видов. Многовидовое слияние приводит к случаям, когда отношения между видами по отдельному гену ( генное дерево) может отличаться от более широкой истории вида ( дерево видов). Это имеет важное значение для теории и практики филогенетика[2][3] и для понимания эволюции генома.
А генное дерево представляет собой двоичный граф, который описывает эволюционные отношения между выборкой последовательностей для нерекомбинирующего локуса. В качестведерево pecies описывает эволюционные отношения между набором видов, предполагая древовидную эволюцию. Однако несколько процессов могут привести к несоответствию между генные деревья и виды деревьев. В Многовидовые сливающиеся Модель обеспечивает основу для вывода филогении видов при учете наследственных полиморфизм и генное дерево-видовое дерево конфликт. Процесс также называют Цензура слитный.[4]
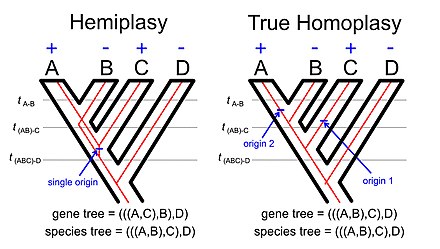
Несоответствие между деревьями генов и деревом видов может привести к случаям, когда символы, которые кажутся гомопластический (приобретаются или теряются независимо в отдельных линиях) с учетом отношений между видами, когда эти признаки фактически имеют одно происхождение; это явление называется гемиплазия.[5] Многие исследования гемиплазии были сосредоточены на геномных характеристиках, таких как нуклеотидные или аминокислотные замены,[6] индели,[7][8] или кариотипические различия[9] (хотя считается, что последний из них менее подвержен гемиплазии, чем многие другие персонажи). Однако фенотипические признаки также могут демонстрировать гемиплазию.[10]
Конгруэнтность генов деревьев и видов деревьев

Если мы рассмотрим корневое дерево из трех таксонов, простейшее нетривиальное филогенетическое дерево, есть три различных топологии дерева.[11] но четыре возможных генных дерева.[12] Существование четырех различных деревьев генов, несмотря на меньшее количество топологий, отражает тот факт, что существуют топологически идентичные деревья генов, которые различаются по времени их объединения. В дереве типа 1 аллели видов A и B сливаются после события видообразования, которое отделяет линию A-B от линии C. В дереве типа 2 аллели видов A и B сливаются перед событием видообразования, которое отделяет линию A-B от линии C (другими словами, дерево типа 2 является глубокая коалесценция дерево). Генные деревья типа 1 и типа 2 совпадают с деревом видов. Два других генных дерева отличаются от дерева видов; два несогласованных генных дерева также глубокая коалесценция деревья.
Распределение времени до слияния фактически непрерывно для всех этих деревьев. Другими словами, точное время слияния любых двух локусов с одним и тем же деревом генов может отличаться. Однако удобно разбивать деревья в зависимости от того, произошло ли слияние до или после самого раннего события видообразования.
Учитывая длину внутренней ветви в сливающихся единицах, легко вычислить вероятность каждого генного дерева.[13] Для диплоидных организмов длина ветви в сливающихся единицах - это количество поколений между событиями видообразования, деленное на удвоенный эффективный размер популяции. Поскольку все три дерева глубокого слияния равновероятны, а два из этих деревьев глубокого слияния противоречат друг другу, легко увидеть, что вероятность того, что корневое дерево генов с тремя таксонами будет конгруэнтно дереву видов, составляет:
Где длина ветви в коалесцентных единицах (Т) также записывается в альтернативной форме: количество поколений (т) деленное на удвоенную эффективную численность популяции (Nе). Памило и Ней[13] также была получена вероятность совпадения для корневых деревьев четырех и пяти таксонов, а также общая верхняя граница вероятности совпадения для более крупных деревьев. Розенберг[14] затем были введены уравнения, используемые для полного набора топологий (хотя большое количество различных филогенетических деревьев становится возможным по мере увеличения количества таксонов[11] делает эти уравнения непрактичными, если количество таксонов не очень ограничено).
Феномен гемиплазии является естественным продолжением основной идеи, лежащей в основе несоответствия между генами и видами деревьев. Если мы рассмотрим распределение некоторого признака, который не соответствует дереву видов, это может отражать гомоплазия (множественное независимое происхождение признака или одно происхождение с последующими множественными потерями) или оно может отражать гемиплазию (единое происхождение признака, связанное с деревом генов, которое не согласуется с деревом видов).
Явление называется неполная сортировка по происхождению (часто сокращенно ILS в научной литературе[7]) связано с явлением. Если мы рассмотрим иллюстрацию гемиплазии с использованием корневого дерева с четырьмя таксонами (см. Изображение справа), линия между общим предком таксонов A, B и C и общим предком таксонов A и B должна быть полиморфной для аллель с производным признаком (например, a сменный элемент вставка[15]) и аллель с наследственным признаком. Концепция неполной сортировки по происхождению в конечном итоге отражает устойчивость полиморфизмов в одном или нескольких событиях видообразования.
Математическое описание многовидового слияния
Плотность вероятности генных деревьев в рамках модели слияния нескольких видов обсуждается вместе с ее использованием для оценки параметров с использованием данных последовательностей с несколькими локусами.
Предположения
Предполагается, что филогения вида известна. Предполагается также полная изоляция после расхождения видов без миграции, гибридизации или интрогрессии. Мы предполагаем отсутствие рекомбинации, так что все сайты в локусе имеют одно и то же дерево генов (топология и время слияния).
Данные и параметры модели
Модель и реализацию этого метода можно применить к любому дереву видов. Например, дерево видов большие обезьяны: человек (ЧАС), шимпанзе (С), горилла (G) и орангутанг (O) считается. Предполагается, что топология дерева видов (((HC) G) O)) известна и зафиксирована в анализе (рисунок 1).[4] Позволять - весь набор данных, где представляют выравнивание последовательностей в локусе , с в общей сложности места.
Размер популяции текущего вида учитывается только в том случае, если от этого вида в некоторых локусах отбирается более одной особи.
Параметры в модели для примера на Рисунке 1 включают три времени расхождения , и и параметры размера популяции для человека; для шимпанзе; и , и для трех видов-предков.
Времена расхождения (s) измеряются ожидаемым числом мутаций на сайт от предкового узла в дереве видов до настоящего времени (рис. 1 из Rannala and Yang, 2003).
Следовательно, параметры .
Вывод на основе правдоподобия
Генеалогия в каждом локусе представлена топологией дерева и время слияния . Данные параметры , распределение вероятностей задается процессом слияния по модели, заданной
Вероятность данных учитывая дерево генов и время слияния (и, следовательно, длину ветвей) в локусе, филогенетическая вероятность Фельзенштейна.[16] Из-за предположения о независимой эволюции по локусам,
К Байесовский вывод на основе совместного условное распределение
Затем апостериорное распределение из дан кем-то
где интеграция представляет собой суммирование по всем возможным топологиям генного дерева и интеграцию за время слияния в каждом локусе.[17]
Распространение генеалогий
Совместное распространение выводится непосредственно в этом разделе. Две последовательности от разных видов могут сливаться только в одну популяцию, которая является предком двух видов. Например, последовательности H и G могут сливаться в популяциях HCG или HCGO, но не в популяциях H или HC. Процессы слияния у разных популяций разные.
Для каждой популяции генеалогия прослеживается назад во времени, до конца популяции во времени. , а количество родословных вход в население и количество родов, выходящих из него записываются. Например, и , для популяции H (таблица 1).[4] Этот процесс называется цензурированный слитный процесс, потому что процесс слияния для одной популяции может быть прекращен до того, как слились все линии, вошедшие в популяцию. Если население состоит из отключенные поддеревья или родословные.
Если одна единица времени определяется как время, необходимое для накопления одной мутации на сайт, любые две линии сливаются со скоростью . Время ожидания до следующего события слияния, которое уменьшает количество родословных от к имеет экспоненциальную плотность
Если , вероятность того, что событие слияния не произойдет между последним и концом популяции в момент времени ; т.е. в течение промежутка времени . Эта вероятность равна и равно 1, если .
(Примечание: следует помнить, что вероятность отсутствия событий в течение интервала времени для пуассоновского процесса со скоростью является . Здесь скорость слияния, когда есть родословная .)
Кроме того, чтобы получить вероятность определенной топологии генного дерева в популяции, если событие слияния происходит в выборке родословных, вероятность того, что конкретная пара родословных сливается, равна .
Умножая эти вероятности вместе, совместное распределение вероятностей топологии генного дерева в популяции и времени его слияния в качестве
- .
Вероятность генного дерева и время слияния для локуса является произведением таких вероятностей для всех популяций. Следовательно, генеалогия на Рисунке 1,[4][18] у нас есть
Влияние на филогенетическую оценку
Слияние многовидовых организмов имеет огромное значение для теории и практики молекулярной филогенетики.[2][3] Поскольку отдельные генные деревья могут отличаться от дерева видов, невозможно оценить дерево для одного локуса и предположить, что дерево генов соответствует дереву видов. Фактически, можно быть практически уверенным, что любое отдельное дерево генов будет отличаться от дерева видов по крайней мере в некоторых отношениях, если рассматривать любое разумное количество таксонов. Однако несоответствие между генами и видами деревьев оказывает влияние на теорию и практику оценки деревьев видов, что выходит за рамки простого наблюдения, что нельзя использовать одно дерево генов для оценки дерева видов, потому что есть часть пространства параметров, где больше всего частое дерево генов несовместимо с деревом видов. Эта часть пространства параметров называется зона аномалии[19] и любые несогласованные генные деревья, которые, как ожидается, будут возникать чаще, чем генное дерево. соответствующие дереву видов называются аномальные генные деревья.
Существование зоны аномалии означает, что нельзя просто оценить большое количество деревьев генов и предположить, что дерево генов, восстановленное наибольшее количество раз, является деревом видов. Конечно, оценка дерева видов путем «демократического голосования» генных деревьев будет работать только для ограниченного числа таксонов за пределами зоны аномалии, учитывая чрезвычайно большое количество возможных филогенетических деревьев.[11] Однако существование аномальных деревьев генов также означает, что простые методы комбинирования деревьев генов, такие как метод расширенного («жадного») консенсуса по правилу большинства или супердерево матричного представления с экономией (MRP)[20][21] подход, не будет последовательный оценщики дерева видов[22][23] (т.е. они будут вводить в заблуждение). Простое создание дерева консенсуса по правилу большинства для деревьев генов, в котором сохраняются группы, которые присутствуют не менее чем в 50% деревьев генов, не будет вводить в заблуждение, пока используется достаточное количество деревьев генов.[22] Однако способность дерева консенсуса по правилам большинства для набора генных деревьев избегать неправильных кладов происходит за счет наличия неразрешенных групп.
Моделирование показало, что есть части пространства параметров дерева видов, где максимальная вероятность оценки филогении - неправильные деревья с возрастающей вероятностью по мере увеличения количества анализируемых данных.[24] Это важно, потому что «подход конкатенации», когда несколько выравниваний последовательностей из разных локусов объединяются, чтобы сформировать одно большое выравнивание суперматрицы, которое затем используется для максимальной вероятности (или Байесовский MCMC) анализ прост в реализации и широко используется в эмпирических исследованиях. Это представляет собой случай неправильной спецификации модели, поскольку конкатенационный подход неявно предполагает, что все генные деревья имеют одинаковую топологию.[25] Действительно, теперь было доказано, что анализ данных, полученных в результате объединения нескольких видов с использованием анализа максимального правдоподобия объединенных данных, не гарантирует сходимость к истинному дереву видов по мере увеличения количества локусов, используемых для анализа.[26][27][28] (т.е. конкатенация максимального правдоподобия статистически несовместима).
Программное обеспечение для вывода с использованием многовидового коалесцента
Существует два основных подхода к филогенетической оценке в рамках многовидовой объединенной структуры: 1) методы полного правдоподобия или полных данных, которые напрямую работают с мультилокусным выравниванием последовательностей, включая как методы максимального правдоподобия, так и байесовские методы, и 2) сводные методы, которые используют сводку исходных данных последовательности, включая двухэтапные методы, которые используют оценочные деревья генов в качестве итоговых входных данных, и SVDQuartets, которые используют счетчики шаблонов сайтов, объединенные по локусам, в качестве суммарных входных данных.
| Программа | Описание | Метод | Рекомендации |
|---|---|---|---|
| АСТРАЛЬНЫЙ | ASTRAL (алгоритм Accurate Species TRee) суммирует набор деревьев генов с использованием метода квартета, генерируя оценку дерева видов с объединяющимися длинами ветвей и поддерживающими значениями (локальные апостериорные вероятности[29]) | Резюме | Мирараб и др. (2014);[30] Zhang et al. (2018)[31] |
| АСТРИД | ASTRID (Accurate Species TRees from Internode Distances) - это расширение метода NJst.[32] ASTRID / NJst - это метод сводного дерева видов, который вычисляет расстояния между узлами на основе набора входных деревьев генов. Метод расстояния вроде присоединение соседа или же минимальная эволюция затем используется для оценки дерева видов с этих расстояний. Обратите внимание, что ASTRID / NJst несовместимы с моделью отсутствующих данных.[33] | Резюме | Вачаспати и Варнов (2015)[34] |
| BPP | Программный пакет Bayesian MCMC для определения филогении и времени расхождения между популяциями в процессе многовидового слияния; также включает метод определения границ видов | Полная вероятность | Ян и др. (2015);[35] Flouri et al. (2018)[36] |
| *ЗВЕРЬ | Программный пакет Bayesian MCMC для определения филогении и времени расхождения среди популяций в процессе многовидового слияния. Реализовано в рамках ЗВЕРЬ программный пакет (произносится Star BEAST) | Полная вероятность | Хелед и Драммонд (2010)[37] |
| MP-EST | Принимает набор деревьев генов в качестве входных данных и генерирует максимальная псевдовероятность оценка породного дерева | Резюме | Лю и др. (2010)[38] |
| СВДКвартет (реализовано в ПАУП *) | PAUP * - это общий пакет филогенетической оценки, который реализует множество методов. SVDquartets - это метод, который показал статистическую согласованность данных, полученных с учетом слияния многовидовых | Метод сайта-шаблона | Чифман и Кубатко (2014)[39] |
Рекомендации
- ^ Дегнан Дж. Х., Розенберг Н. А. (июнь 2009 г.). «Несоответствие дерева генов, филогенетический вывод и слияние многовидовых». Тенденции в экологии и эволюции. 24 (6): 332–40. Дои:10.1016 / j.tree.2009.01.009. PMID 19307040.
- ^ а б Мэддисон WP (1997-09-01). «Генные деревья в видовых деревьях». Систематическая биология. 46 (3): 523–536. Дои:10.1093 / sysbio / 46.3.523. ISSN 1063-5157.
- ^ а б Эдвардс С.В. (январь 2009 г.). «Возникает ли новая общая теория молекулярной систематики?». Эволюция; Международный журнал органической эволюции. 63 (1): 1–19. Дои:10.1111 / j.1558-5646.2008.00549.x. PMID 19146594.
- ^ а б c d Раннала Б., Ян З. (август 2003 г.). «Байесовская оценка времени расхождения видов и размеров предковой популяции с использованием последовательностей ДНК из нескольких локусов». Генетика. 164 (4): 1645–56. ЧВК 1462670. PMID 12930768.
- ^ Avise JC, Робинсон TJ (июнь 2008 г.). Кубатко Л (ред.). «Гемиплазия: новый термин в лексиконе филогенетики». Систематическая биология. 57 (3): 503–7. Дои:10.1080/10635150802164587. PMID 18570042.
- ^ Мендес Ф.К., Хан Й., Хан М.В. (декабрь 2016 г.). «Несоответствие дерева генов может порождать закономерности уменьшения сходимости с течением времени». Молекулярная биология и эволюция. 33 (12): 3299–3307. Дои:10.1093 / molbev / msw197. PMID 27634870.
- ^ а б Джарвис Э.Д., Мирараб С., Аберер А.Дж., Ли Б., Хоуд П., Ли С. и др. (Декабрь 2014 г.). «Полногеномный анализ позволяет выявить ранние ветви на древе жизни современных птиц». Наука. 346 (6215): 1320–31. Дои:10.1126 / science.1253451. ЧВК 4405904. PMID 25504713.
- ^ Хоуде П., Браун Е.Л., Нарула Н., Минджарес Ю., Мирараб С. (2019-07-06). «Филогенетический сигнал инделей и неоавианское излучение». Разнообразие. 11 (7): 108. Дои:10.3390 / d11070108.
- ^ Робинсон Т.Дж., Руис-Эррера А., Ависе ЮК (сентябрь 2008 г.). «Гемиплазия и гомоплазия в кариотипической филогенезе млекопитающих». Труды Национальной академии наук Соединенных Штатов Америки. 105 (38): 14477–81. Дои:10.1073 / pnas.0807433105. ЧВК 2567171. PMID 18787123.
- ^ Герреро РФ, Хан МВТ (декабрь 2018 г.). «Количественная оценка риска гемиплазии в филогенетическом заключении». Труды Национальной академии наук Соединенных Штатов Америки. 115 (50): 12787–12792. Дои:10.1073 / pnas.1811268115. ЧВК 6294915. PMID 30482861.
- ^ а б c Фельзенштейн Дж (март 1978 г.). «Число эволюционных деревьев». Систематическая зоология. 27 (1): 27. Дои:10.2307/2412810.
- ^ Hobolth A, Christensen OF, Mailund T, Schierup MH (февраль 2007 г.). «Геномные отношения и время видообразования человека, шимпанзе и гориллы, выведенные из объединенной скрытой модели Маркова». PLoS Genetics. 3 (2): e7. Дои:10.1371 / journal.pgen.0030007. ЧВК 1802818. PMID 17319744.
- ^ а б Памило П., Ней М. (сентябрь 1988 г.). «Взаимосвязь между деревьями генов и деревьями видов». Молекулярная биология и эволюция. 5 (5): 568–83. Дои:10.1093 / oxfordjournals.molbev.a040517. PMID 3193878.
- ^ Розенберг Н.А. (март 2002 г.). «Вероятность топологического соответствия деревьев генов и деревьев видов». Теоретическая популяционная биология. 61 (2): 225–47. Дои:10.1006 / tpbi.2001.1568. PMID 11969392.
- ^ Сух А, Смедс Л., Эллегрен Х (август 2015 г.). Пенни Д. (ред.). «Динамика неполной сортировки по древнему адаптивному излучению неоавийских птиц». PLoS Биология. 13 (8): e1002224. Дои:10.1371 / journal.pbio.1002224. ЧВК 4540587. PMID 26284513.
- ^ Фельзенштейн Дж (1981).«Эволюционные деревья из последовательностей ДНК: подход максимального правдоподобия». Журнал молекулярной эволюции. 17 (6): 368–76. Дои:10.1007 / BF01734359. PMID 7288891.
- ^ Сюй Б, Ян З (декабрь 2016 г.). «Проблемы оценки деревьев видов в рамках модели слияния нескольких видов». Генетика. 204 (4): 1353–1368. Дои:10.1534 / генетика.116.190173. ЧВК 5161269. PMID 27927902.
- ^ Ян З (2014). Молекулярная эволюция: статистический подход (Первое изд.). Оксфорд: Издательство Оксфордского университета. С. Глава 9. ISBN 9780199602605. OCLC 869346345.
- ^ Дегнан Дж. Х., Розенберг Н. А. (май 2006 г.). Уэйкли Дж (ред.). «Несоответствие видов деревьев их наиболее вероятным генным деревьям». PLoS Genetics. 2 (5): e68. Дои:10.1371 / journal.pgen.0020068. ЧВК 1464820. PMID 16733550.
- ^ Баум Б.Р. (февраль 1992 г.). «Объединение деревьев как способ объединения наборов данных для филогенетических выводов и желательность объединения генных деревьев». ТАКСОН. 41 (1): 3–10. Дои:10.2307/1222480. ISSN 0040-0262.
- ^ Раган М.А. (март 1992 г.). «Филогенетический вывод на основе матричного представления деревьев». Молекулярная филогенетика и эволюция. 1 (1): 53–58. Дои:10.1016 / 1055-7903 (92) 90035-Ф.
- ^ а б Дегнан Дж. Х., Де Джорджио М, Брайант Д., Розенберг Н. А. (февраль 2009 г.). «Свойства методов консенсуса для вывода деревьев видов из деревьев генов». Систематическая биология. 58 (1): 35–54. Дои:10.1093 / sysbio / syp008. ЧВК 2909780. PMID 20525567.
- ^ Ван И, Дегнан Дж.Х. (02.05.2011). «Выполнение матричного представления с экономией для вывода видов из генных деревьев». Статистические приложения в генетике и молекулярной биологии. 10 (1). Дои:10.2202/1544-6115.1611.
- ^ Кубатко Л.С., Дегнан Дж.Х. (февраль 2007 г.). Коллинз Т. (ред.). «Несоответствие филогенетических оценок от объединенных данных при слиянии». Систематическая биология. 56 (1): 17–24. Дои:10.1080/10635150601146041. PMID 17366134.
- ^ Warnow T (май 2015 г.). «Конкатенационный анализ при неполной сортировке по происхождению». PLoS токи. 7. Дои:10.1371 / currents.tol.8d41ac0f13d1abedf4c4a59f5d17b1f7. ЧВК 4450984. PMID 26064786.
- ^ Рох С., Сталь М (март 2015 г.). «Реконструкция дерева на основе правдоподобия при объединении наборов данных выровненных последовательностей может быть статистически несовместимой». Теоретическая популяционная биология. 100C: 56–62. Дои:10.1016 / j.tpb.2014.12.005. PMID 25545843.
- ^ Мендес Ф.К., Хан М.В. (январь 2018 г.). «Почему конкатенация не удается вблизи зоны аномалии». Систематическая биология. 67 (1): 158–169. Дои:10.1093 / sysbio / syx063. PMID 28973673.
- ^ Рох С., Нут М., Варнов Т. (март 2019 г.). Кубатко Л (ред.). «Притяжение длинных ветвей в оценке дерева видов: несогласованность методов разделенного правдоподобия и сводных методов на основе топологии». Систематическая биология. 68 (2): 281–297. Дои:10.1093 / sysbio / syy061. PMID 30247732.
- ^ Сайяри Э., Мирараб С. (июль 2016 г.). «Быстрое вычисление поддержки локальных ветвей на основе частот квартета». Молекулярная биология и эволюция. 33 (7): 1654–68. Дои:10.1093 / molbev / msw079. ЧВК 4915361. PMID 27189547.
- ^ Мирараб С., Реаз Р., Байзид М.С., Циммерманн Т., Свенсон М.С., Варнов Т. (сентябрь 2014 г.). «ASTRAL: оценка деревьев видов на основе генома». Биоинформатика. 30 (17): i541-8. Дои:10.1093 / биоинформатика / btu462. ЧВК 4147915. PMID 25161245.
- ^ Чжан С., Раби М., Сайяри Е., Мирараб С. (май 2018 г.). «ASTRAL-III: полиномиальная временная реконструкция дерева видов из частично разрешенных деревьев генов». BMC Bioinformatics. 19 (Дополнение 6): 153. Дои:10.1186 / s12859-018-2129-у. ЧВК 5998893. PMID 29745866.
- ^ Лю, Лян; Ю, Лили (01.10.2011). "Оценка видовых деревьев по неукорененным генным деревьям". Систематическая биология. 60 (5): 661–667. Дои:10.1093 / sysbio / syr027. ISSN 1076-836X.
- ^ Родос Дж. А., Нут М. Г., Варнов Т. (январь 2020 г.). «NJst и ASTRID статистически несовместимы при случайной модели отсутствующих данных». arXiv: 2001.07844 https://arxiv.org/abs/2001.07844
- ^ Вачаспати, Пранджал; Варнов, Тэнди (декабрь 2015 г.). "ASTRID: Точные исследования видов с расстояний между узлами". BMC Genomics. 16 (S10): S3. Дои:10.1186 / 1471-2164-16-S10-S3. ISSN 1471-2164. ЧВК 4602181. PMID 26449326.
- ^ Ян З (01.10.2015). «Программа BPP для оценки древесных пород и определения границ пород». Современная зоология. 61 (5): 854–865. Дои:10.1093 / czoolo / 61.5.854. ISSN 2396-9814.
- ^ Флури Т., Цзяо X, Раннала Б., Ян Зи (октябрь 2018 г.). Йодер А.Д. (ред.). «Вывод дерева видов с помощью BPP с использованием геномных последовательностей и многовидового слияния». Молекулярная биология и эволюция. 35 (10): 2585–2593. Дои:10.1093 / molbev / msy147. ЧВК 6188564. PMID 30053098.
- ^ Heled, J .; Драммонд, А. Дж. (01.03.2010). "Байесовский вывод видовых деревьев из многолокационных данных". Молекулярная биология и эволюция. 27 (3): 570–580. Дои:10.1093 / молбев / msp274. ISSN 0737-4038. ЧВК 2822290. PMID 19906793.
- ^ Лю Л., Ю. Л., Эдвардс С.В. (октябрь 2010 г.). «Подход максимального псевдо-правдоподобия для оценки деревьев видов в рамках модели слияния». BMC Эволюционная биология. 10 (1): 302. Дои:10.1186/1471-2148-10-302. ЧВК 2976751. PMID 20937096.
- ^ Чифман Дж., Кубатко Л (декабрь 2014 г.). «Квартетный вывод из данных SNP в рамках объединенной модели». Биоинформатика. 30 (23): 3317–24. Дои:10.1093 / биоинформатика / btu530. ЧВК 4296144. PMID 25104814.








































![{ Displaystyle ехр {- { гидроразрыва {п (п-1)} { тета}} [ тау - (т_ {м} + т_ {м-1} + ldots + т_ {п + 1} )]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/2f435c3e48b6ad522af7d79165c8713ab6a42446)







![{ displaystyle prod _ {j = n + 1} ^ {m} { Big [} { frac {2} { theta}} exp { Big {} - { frac {j (j- 1)} { theta}} t_ {j} { Big }} { Big]} exp { Big {} - { frac {n (n-1)} { theta}} ( тау - (t_ {m} + t_ {m + 1} + ldots + t_ {n + 1})) { Big }}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/a7937f1541db74c1d61321eb52a49d5a32f19407)
![{ Displaystyle { begin {выровнено} е (G_ {i} mid Theta) & = [2 / theta _ {H} exp {- 6t_ {3} ^ {(H)} / theta _ {H} } exp {- 2 ( tau _ {HC} -t_ {3} ^ {(H)}) / theta _ {H} }] & {} times [2 / theta _ {C} exp {- 2t_ {2} ^ {(C)} / theta _ {C} }] & {} times [2 / theta _ {HC} exp {-6t_ {3} ^ {HC} / theta _ {HC} }] times [2 / theta _ {HC} exp {- 2t_ {2} ^ {HC} / theta _ {HC } }] & {} times [ exp {- 2 ( tau _ {HCG} - tau _ {HG} - (t_ {3} ^ {HC} + t_ {2} ^ {HC })) / theta _ {HCG} }] & {} times [2 / theta _ {HCGO} exp {- 6t_ {3} ^ {HCGO} / theta _ {HCGO} }] times [2 / theta _ {HCGO} exp {- 2t_ {2} ^ {HCGO} / theta _ {HCGO} }] end {выровнено}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/2d396905de66e7b66c05298abe56ddc5be144a53)