WikiDer > WPGMA
WPGMA (Wвосьмой пвоздуха граммгруппа Mэтод с Аrithmetic Mean) представляет собой простую агломерацию (снизу вверх) иерархическая кластеризация метод, обычно относящийся к Сокаль и Michener.[1]
Метод WPGMA аналогичен его невзвешенный вариант, UPGMA метод.
Алгоритм
Алгоритм WPGMA строит корневое дерево (дендрограмма), который отражает структуру, присутствующую в попарном матрица расстояний (или матрица сходства). На каждом шаге ближайшие два кластера, скажем, и , объединяются в кластер более высокого уровня . Затем его расстояние до другого кластера это просто среднее арифметическое средних расстояний между членами и и и :
Алгоритм WPGMA создает корневые дендрограммы и требует допущения о постоянной скорости: он дает ультраметрический дерево, в котором расстояния от корня до всех концов веток равны. Этот ультраметричность предположение называется молекулярные часы когда советы включают ДНК, РНК и белок данные.
Рабочий пример
Этот рабочий пример основан на JC69 матрица генетических расстояний, вычисленная из 5S рибосомальная РНК выравнивание последовательностей пяти бактерий: Bacillus subtilis (), Bacillus stearothermophilus (), Лактобациллы viridescens (), Ахолеплазма хоть (), и Micrococcus luteus ().[2][3]
Первый шаг
- Первая кластеризация
Предположим, что у нас есть пять элементов и следующая матрица попарных расстояний между ними:
| а | б | c | d | е | |
|---|---|---|---|---|---|
| а | 0 | 17 | 21 | 31 | 23 |
| б | 17 | 0 | 30 | 34 | 21 |
| c | 21 | 30 | 0 | 28 | 39 |
| d | 31 | 34 | 28 | 0 | 43 |
| е | 23 | 21 | 39 | 43 | 0 |
В этом примере это наименьшее значение , поэтому мы соединяем элементы и .
- Оценка длины первой ветви
Позволять обозначим узел, к которому и теперь подключены. Параметр гарантирует, что элементы и равноудалены от . Это соответствует ожиданиям ультраметричность гипотеза. и к тогда имейте длину (см. финальную дендрограмму)
- Первое обновление матрицы расстояний
Затем мы приступаем к обновлению исходной матрицы расстояний в новую матрицу расстояний (см. ниже), уменьшенного в размере на одну строку и один столбец из-за кластеризации с Значения жирным шрифтом в соответствуют новым расстояниям, рассчитанным по усреднение расстояний между каждым элементом первого кластера и каждый из оставшихся элементов:
Значения, выделенные курсивом в не затрагиваются обновлением матрицы, поскольку они соответствуют расстояниям между элементами, не участвующими в первом кластере.
Второй шаг
- Вторая кластеризация
Теперь мы повторяем три предыдущих шага, начиная с новой матрицы расстояний. :
| (а, б) | c | d | е | |
|---|---|---|---|---|
| (а, б) | 0 | 25.5 | 32.5 | 22 |
| c | 25.5 | 0 | 28 | 39 |
| d | 32.5 | 28 | 0 | 43 |
| е | 22 | 39 | 43 | 0 |
Здесь, это наименьшее значение , поэтому мы присоединяемся к кластеру и элемент .
- Оценка длины второй ветви
Позволять обозначим узел, к которому и теперь подключены. Из-за ограничения ультраметричности ветви, соединяющиеся или же к , и к равны и имеют следующую длину:
Вычисляем недостающую длину ветки: (см. финальную дендрограмму)
- Обновление матрицы второго расстояния
Затем мы приступаем к обновлению матрицу в новую матрицу расстояний (см. ниже), уменьшенного в размере на одну строку и один столбец из-за кластеризации с :
Следует отметить, что это средний расчет нового расстояния не учитывает больший размер кластер (два элемента) относительно (один элемент). По аналогии:
Поэтому процедура усреднения придает дифференциальный вес начальным расстояниям матрицы . Это причина, по которой метод взвешенныйне по математической процедуре, а по отношению к начальным расстояниям.
Третий шаг
- Третья кластеризация
Мы снова повторяем три предыдущих шага, начиная с обновленной матрицы расстояний. .
| ((а, б), д) | c | d | |
|---|---|---|---|
| ((а, б), д) | 0 | 32.25 | 37.75 |
| c | 32.25 | 0 | 28 |
| d | 37.75 | 28 | 0 |
Здесь, это наименьшее значение , поэтому мы соединяем элементы и .
- Оценка длины третьей ветви
Позволять обозначим узел, к которому и подключены. и к тогда имейте длину (см. финальную дендрограмму)
- Обновление третьей матрицы расстояний
Необходимо обновить одну запись:
Заключительный этап
Финал матрица:
| ((а, б), д) | (CD) | |
|---|---|---|
| ((а, б), д) | 0 | 35 |
| (CD) | 35 | 0 |
Итак, мы присоединяемся к кластерам и .
Позволять обозначают (корневой) узел, к которому и подключены. и к тогда имейте длины:
Вычисляем две оставшиеся длины ветвей:
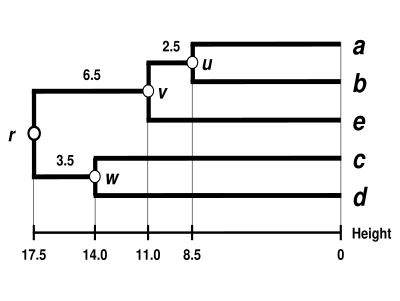
Дендрограмма WPGMA
Дендрограмма завершена. Он ультраметрический, потому что все наконечники ( к ) равноудалены от :
Таким образом, дендрограмма основана на , его самый глубокий узел.
Сравнение с другими связями
Альтернативные схемы связи включают однократная кластеризация, полная кластеризация связей, и Кластеризация средних связей UPGMA. Реализация другой связи - это просто вопрос использования другой формулы для расчета межкластерных расстояний на этапах обновления матрицы расстояний в вышеупомянутом алгоритме. Полная кластеризация связей позволяет избежать недостатка альтернативного метода кластеризации одиночных связей - так называемого явление сцепления, где кластеры, сформированные посредством кластеризации с одной связью, могут быть принудительно объединены из-за того, что отдельные элементы находятся близко друг к другу, даже если многие элементы в каждом кластере могут быть очень удалены друг от друга. Полная связь имеет тенденцию находить компактные группы приблизительно равного диаметра.[4]
 | |||
| Односвязная кластеризация. | Кластеризация с полной связью. | Средняя кластеризация связей: WPGMA. | Кластеризация средней связи: UPGMA. |
Смотрите также
- Соседство
- Молекулярные часы
- Кластерный анализ
- Односвязная кластеризация
- Кластеризация с полной связью
- Иерархическая кластеризация
Рекомендации
- ^ Сокаль, Michener (1958). «Статистический метод оценки систематических взаимосвязей». Бюллетень науки Канзасского университета. 38: 1409–1438.
- ^ Эрдманн В.А., Вольтерс Дж. (1986). «Коллекция опубликованных последовательностей рибосомных РНК 5S, 5.8S и 4.5S». Исследования нуклеиновых кислот. 14 Suppl (Suppl): r1–59. Дои:10.1093 / nar / 14.suppl.r1. ЧВК 341310. PMID 2422630.
- ^ Olsen GJ (1988). «Филогенетический анализ с использованием рибосомальной РНК». Методы в энзимологии. 164: 793–812. Дои:10.1016 / с0076-6879 (88) 64084-5. PMID 3241556.
- ^ Everitt, B.S .; Ландау, С .; Лиз, М. (2001). Кластерный анализ. 4-е издание. Лондон: Арнольд. п. 62–64.