WikiDer > CAPP-Seq
| Акроним | CAPP-Seq |
|---|---|
| Использует | Количественная оценка низкого уровня ctDNA от онкологических больных. |
| Известные эксперименты | CAPP-Seq применялся при немелкоклеточном раке легкого (НМРЛ) для выявления рецидивирующих соматических изменений ctDNA. |
| Похожие материалы | Бесклеточная опухолевая ДНК |
CANcer пперсонализированный ппрофилирование глубоким Seqучастие (CAPP-Seq) - это метод секвенирования нового поколения, используемый для количественного определения циркулирующей ДНК в рак (втДНК). Метод был представлен в 2014 году лабораториями Эша Ализаде и Максимилиана Дина в Стэнфорде в качестве инструмента для измерения Внеклеточная опухолевая ДНК который высвобождается из мертвых опухолевых клеток в кровь и, таким образом, может отражать весь геном опухоли. Этот метод можно обобщить для любого типа рака, который, как известно, имеет повторяющиеся мутации.[1] CAPP-Seq может обнаруживать одну молекулу мутантной ДНК в 10 000 молекул здоровой ДНК. Оригинальный метод[1] В 2016 году была доработана для сверхчувствительного обнаружения за счет интеграции нескольких стратегий подавления ошибок, получивших название интегрированного цифрового подавления ошибок (iDES).[2] Использование цтДНК в этом методе не следует путать с циркулирующими опухолевыми клетками (ЦКО); это две разные сущности.[3]
Первоначально описывался как метод обнаружения и мониторинга рака легких,[1][2] CAPP-Seq был успешно адаптирован для лечения широкого спектра раковых заболеваний несколькими независимыми группами. К ним относятся диффузная В-клеточная лимфома большого размера (DLBCL),[4] фолликулярная лимфома (FL),[4] посттрансплантационное лимфопролиферативное заболевание (PTLD),[5] метастатический колоректальный рак к яичнику,[6] рак пищевода,[7] панкреатический рак,[8] Рак мочевого пузыря,[9] лейомиосаркома,[10] разнообразный взрослый и детский саркомы,[11] среди прочего.
Метод
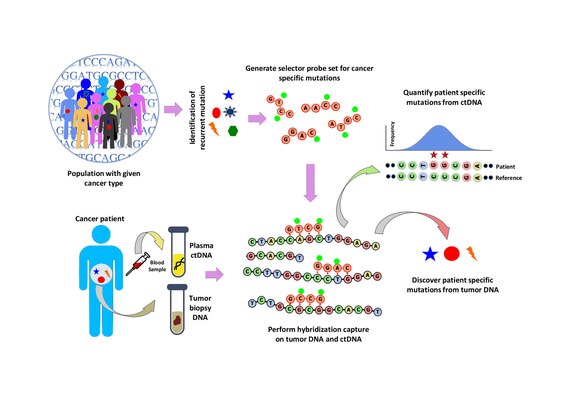
Популяционный анализ проводится для выявления повторяющихся мутаций при данном типе рака. Это делается путем анализа общедоступных наборов данных, таких как База данных рака COSMIC и TCGA. Разработан "селектор", который состоит из биотинилированный ДНК-олигонуклеотидные зонды, нацеленные на рекуррентно мутировавшие области, выбранные для конкретного типа рака. Селектор выбирается с использованием подхода многофазной биоинформатики. С помощью селектора выполняется гибридизационный захват на основе зонда на опухолевой и нормальной ДНК для обнаружения мутаций, специфичных для пациента. Затем гибридизационный захват также применяется к цДНК для количественной оценки мутаций, которые были обнаружены ранее.[1]
извлечение ктДНК и подготовка библиотеки
У пациентов собирают периферическую кровь и выделяют цДНК из ≥1 мл плазма. Входная ДНК может составлять всего 4 нг.[1]
При адаптации этого протокола для работы с втДНК было четыре основных цели:
- 1) для оптимизации эффективности лигирования адаптера
- 2) для уменьшения количества циклов ПЦР, необходимых после лигирования
- 3) для сохранения естественного распределения ктДНК по размерам (в среднем 170 бит / с)
- 4) минимизировать вариабельность глубины охвата последовательностей во всех захваченных регионах.
Это было достигнуто путем проведения лигирования адаптера при 16 ℃ в течение 16 часов для повышения эффективности лигирования адаптера и восстановления. Наиболее важная адаптация происходит во время ферментативных стадий и стадий очистки; они выполняются с шариком, чтобы свести к минимуму этапы переноса пробирки, что увеличивает извлечение.
Дизайн селектора
В CAPP-Seq разработка селектора является важным шагом, который определяет повторяющиеся мутации в конкретном типе рака с использованием общедоступных данных секвенирования следующего поколения. Для включения в селектор CAPP-seq повторяющиеся мутации, которые обогащаются в популяции, описываются индексом - Индекс повторения (RI). RI - это количество мутаций на килобазу данного геномного локуса пациента, несущего определенные мутации. RI представляет собой частоту рецидивов на уровне пациента, оцененную для соматических мутаций и всех мутаций. Известные и ведущие повторяющиеся мутации в популяции могут быть ранжированы на основе RI, и поэтому RI используется для разработки селектора. Для разработки селектора используется шестиэтапная стратегия проектирования.[1]
- Этап 1. Выявление часто мутирующих мутаций известных драйверов с использованием общедоступных данных.
- Фаза 2: максимальный охват SNV среди пациентов был определен путем ранжирования их экзонных RI.
- Фаза 3 и 4: были отобраны экзоны с более высоким RI.
- Фаза 5: Добавление ранее предсказанных мутаций драйверов.
- Фаза 6: добавление реорганизации повторяющихся слияний генов, специфичных для конкретного рака.
Рак человека неоднороден, и рецидивирующие мутации рака присутствуют только у меньшинства пациентов. Следовательно, тщательная и неизбыточная конструкция селектора является жизненно важной частью CAPP-Seq, а также размер селектора связан с его последующими затратами.
Захват гибридизации и секвенирование
Эта секция не цитировать любой источники. (Январь 2020) (Узнайте, как и когда удалить этот шаблон сообщения) |
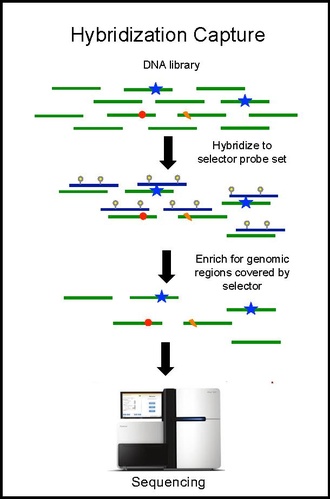
Гибридизационный захват с помощью набора селекторных зондов выполняется на опухолевой ДНК из биопсии и секвенирован на глубину ~ 10 000-кратного покрытия. Биотинилированные селекторные зонды селективно связываются с участками библиотеки ДНК, которые были выбраны так, чтобы в них повторялись мутации при данном типе рака. Таким образом, у вас остаётся библиотека меньшего размера, которая обогащена только для желаемых регионов, которые затем можно секвенировать. Это позволяет определять специфические мутации пациента. Затем проводят гибридизационный захват с тем же селектором на ктДНК из крови для количественной оценки ранее идентифицированных мутаций у пациента. CAPP-Seq можно применять к цТДНК из нескольких образцов крови в разные моменты времени, чтобы проследить эволюцию опухоли.
Вычислительный конвейер для CAPP-seq
В анализ данных CAPP-Seq входит ряд шагов, от обнаружения мутации до проверки, и программное обеспечение с открытым исходным кодом может выполнять большую часть анализа. После первого шага варианта вызова зародышевые линии и потеря гетерозиготности (LOH) мутации удаляются в CAPP-seq, чтобы уменьшить фоновые ошибки. Несколько тестов статистической значимости могут быть выполнены на фоне для всех типов вызова вариантов. Например, статистическая значимость SNV, полученных из опухоли, может быть оценена путем случайной выборки фоновых аллелей с использованием Метод Монте-Карло. Для вызовов indel статистическая значимость рассчитывается с применением отдельного метода, в котором использовался анализ, специфичный для цепочки, путем Z-тест показано в предыдущей работе.[1] Наконец, этапы вычислительной проверки сокращают количество ложных срабатываний. Однако надежная вычислительная среда, специфичная для анализа данных CAPP-seq, очень востребована в этой области.
Чувствительность
Чувствительность этой технологии зависит от эффективной конструкции селектора и сильно зависит от размера когорты и типа исследуемого рака. Отсутствие фона для поиска статистически значимых повторяющихся вариантов ограничивает его производительность из-за стохастического шума и биологической изменчивости. Рабочая характеристика приемника (ROC) анализ нескольких больных раком и пациентов, излечившихся от рака (образец, собранный на разных стадиях опухоли, момент времени циркуляции ДНК, лечение и т. Д.), Показал, что CAPP-seq имеет более высокую чувствительность и специфичность по сравнению с предыдущими методами для немелкоклеточных рак легких.[1]
Ограничения
На предел обнаружения CAPP-Seq влияют три основных фактора: входное количество молекул цтДНК, перекрестное загрязнение образца, потенциальное аллельное смещение в реагенте захвата и ПЦР или ошибки секвенирования. ктДНК может быть обнаружена на нижнем пределе 0,025% фракционного содержания в крови. Было обнаружено, что перекрестное загрязнение образцов вносит очень небольшой вклад, и отчеты показали минимальное аллельное смещение в сторону захвата референсных аллелей в PBL (лимфоциты периферической крови). Ошибки ПЦР и секвенирования также минимальны.[1] Этот метод становится сомнительным, если цтДНК присутствует на низких уровнях 0,01%. Кроме того, когда наблюдается меньшее выделение цтДНК из-за стабильности роста опухоли при терапии, обнаружение затрудняется.[12][13]
Высвобождается ли ктДНК с одинаковой или неравной скоростью из первичных опухолей и метастатические заболевания пока неизвестно. Этот факт следует учитывать при выполнении CAPP-Seq, поскольку он может вызвать проблемы при определении опухолевой нагрузки и клональной эволюции, если разные опухоли или клоны отмирают и высвобождают свою ДНК с разной скоростью. Также неизвестно, как гистология опухоли влияет на высвобождение ктДНК.[1]
Еще одно серьезное ограничение при использовании только уровней ctDNA для определения опухолевой нагрузки заключается в том, что ctDNA может предсказывать только остаточную опухоль, она ничего не может сказать о локализации опухоли. Это означает, что CAPP-Seq может лучше всего использоваться в дополнение к другим подходам к секвенированию.[10] для визуализации бремени болезни в разное время. Таким образом, техническая чувствительность, воспроизводимость, специфичность и потребность в экспертных знаниях для анализа большого количества данных - вот некоторые из проблем, связанных с этим методом.
Преимущества
CAPP-Seq имеет много преимуществ перед другими методами, такими как цифровая полимеразная цепная реакция (dPCR) и ампликон последовательность действий. CAPP-Seq может исследовать множество локусов в одном эксперименте по сравнению с dPCR и секвенированием ампликона, которые используют несколько разных экспериментов и, следовательно, используют гораздо больше образца. Еще одно преимущество состоит в том, что CAPP-Seq может не только обнаруживать точечные мутации, но и обнаруживать инделы, структурные вариации, и варианты числа копий[14] а также помогает контролировать минимальную остаточную болезнь.[15]
Еще одно преимущество CAPP-Seq заключается в том, что, поскольку он нацелен только на определенные интересующие области генома, он более экономичен, чем весь секвенирование экзома и секвенирование всего генома которые на 171X и 44X дороже соответственно.[1] Кроме того, нет необходимости в дискретной оптимизации для отдельных пациентов.
Использование циркулирующей опухолевой ДНК, в отличие от биопсии солидных опухолей, позволяет анализировать полный репертуар опухолевых клеток, рассредоточенных по опухоли и отдаленных метастазах. Следовательно, есть больше шансов найти все мутации, связанные с этим раком. Наличие полного обзора рака и его движущих сил позволит улучшить планы лечения и ведения болезни.
Приложения
Мониторинг опухолевой нагрузки
При лечении рака полезно иметь точные измерения общей нагрузки на организм. Это помогает определить прогностическую значимость и реакцию на лечение. Обычно это делается с помощью компьютерной томографии (Компьютерная томография), позитронно-эмиссионная томография (ПЭТ сканирование) или магнитно-резонансной томографии (МРТ).[16] Эти процедуры медицинской визуализации дороги и не лишены собственных проблем. Эти методы визуализации не позволяют точно разрешить небольшие опухоли (≤1 см в диаметре).[14] На визуализацию также могут влиять радиационно-индуцированное воспаление и фиброзные изменения, что затрудняет определение наличия остаточной опухоли или просто эффектов лечения.[1]
Было обнаружено, что уровни цтДНК в плазме значительно коррелируют с объемом опухоли по сравнению с результатами медицинской визуализации (КТ, ПЭТ и МРТ).[1][14][17][18] Обнаружение ctDNA может предсказать остаточную опухоль или неминуемый рецидив, в некоторых случаях даже лучше, чем медицинская визуализация и современные методы.
Прогностический индикатор
На сегодняшний день обнаружение ctDNA является предиктором рецидива во многих исследованиях. В исследовании [14] на поздней стадии НМРЛ (немелкоклеточный рак легкого) они обнаружили два случая, когда втДНК правильно определила исход болезни пациента, когда медицинская визуализация была неправильной. В одном случае визуализация предсказывала рецидив на основании подозреваемой остаточной опухоли, которая оказалась только вызванной радиацией воспалением, но цтДНК не была обнаружена, и у пациента не было рецидива. В другом случае визуализация не показала опухоли, но была обнаружена цтДНК, и вскоре после этого у пациента случился рецидив. В другом исследовании [18] на DLBCL (диффузная крупноклеточная В-клеточная лимфома), цтДНК также оказалась предиктором рецидива.
Генотипирование опухоли без биопсии
Биопсия инвазивна и связана с риском для пациента. Следовательно, многократные биопсии для отслеживания прогрессирования заболевания являются редкостью, и диагностические биопсии используются для получения генетической информации. Это может быть проблематично из-за неоднородности опухоли и ее эволюции. Во-первых, при биопсии отбирается только одна часть опухоли, и поскольку опухоли неоднородны, они не охватывают весь генетический ландшафт опухоли. Во-вторых, после лечения опухоли развиваются, и могут появиться новые мутации, не представленные в диагностической выборке.[1][14]
Генотипирование опухоли без биопсии с помощью CAPP-Seq и ctDNA решает многие из этих проблем. Простой анализ крови неинвазивен, гораздо безопаснее и легче подвергать онкологических больных многократному анализу в течение курса лечения. Использование ctDNA дает лучший образец опухолевой ДНК по сравнению с одной областью опухоли, собранной при биопсии, что позволяет лучше оценить гетерогенность опухоли. Взятие нескольких образцов ктДНК в разные моменты времени после курса лечения позволяет выявить эволюцию опухоли. Это может помочь обнаружить появление мутаций, которые придают устойчивость к целевой терапии, и позволить соответствующим образом скорректировать курс лечения. CAPP-Seq, в частности, позволяет проводить скрининг нескольких геномных участков, что станет важным, поскольку список раковых мутаций, важных для лечения, продолжает расти.[14] В исследовании[1] для поздней стадии НМРЛ они выполнили версию CAPP-Seq, в которой биопсия опухоли не была секвенирована первой, и они смогли правильно классифицировать 100% образцов плазмы пациентов с 0% ложноположительной частотой. Это показывает, что даже без предварительного знания опухолевых мутаций их можно точно обнаружить только с помощью цтДНК.
Рекомендации
- ^ а б c d е ж грамм час я j k л м п о Ньюман А.М., Братман С.В., То Дж., Винн Дж. Ф., Эклов Н. С., Модлин Л. А. и др. (Май 2014 г.). «Сверхчувствительный метод количественного определения циркулирующей опухолевой ДНК с широким охватом пациентов». Природа Медицина. 20 (5): 548–54. Дои:10,1038 / нм.3519. ЧВК 4016134. PMID 24705333.
- ^ а б Ньюман А.М., Лавджой А.Ф., Класс Д.М., Курц Д.М., Чабон Дж. Дж., Шерер Ф. и др. (Май 2016). «Встроенное подавление цифровых ошибок для улучшенного обнаружения циркулирующей ДНК опухоли». Природа Биотехнологии. 34 (5): 547–555. Дои:10.1038 / nbt.3520. ЧВК 4907374. PMID 27018799.
- ^ Бетеговда К., Саусен М., Лири Р.Дж., Кинде И., Ван И, Агравал Н. и др. (Февраль 2014). «Обнаружение циркулирующей опухолевой ДНК на ранних и поздних стадиях злокачественных новообразований человека». Научная трансляционная медицина. 6 (224): 224ra24. Дои:10.1126 / scitranslmed.3007094. ЧВК 4017867. PMID 24553385.
- ^ а б Шерер Ф., Курц Д.М., Ньюман А.М., Штер Х., Крейг А.Ф., Исфахани М.С. и др. (Ноябрь 2016 г.). «Отличительные биологические подтипы и паттерны эволюции генома лимфомы, выявленные циркулирующей опухолевой ДНК». Научная трансляционная медицина. 8 (364): 364ra155. Дои:10.1126 / scitranslmed.aai8545. ЧВК 5490494. PMID 27831904.
- ^ Су Дж., Шроерс-Мартин Дж., Гарофало А., Курц Д., Д'Эмилио Н., Луикарт Х., Ализаде А., Хуш К. (20 мая 2018 г.). «Раннее выявление посттрансплантационного лимфопролиферативного расстройства с использованием циркулирующей опухолевой ДНК». Журнал клинической онкологии. 36 (15_suppl): 7572. Дои:10.1200 / JCO.2018.36.15_suppl.7572.
- ^ Ивахаси Н., Сакаи К., Ногучи Т., Яхата Т., Тодзима С., Нишио К., Ино К. (ноябрь 2018 г.). «Комплексный анализ мутаций генов в жидких биоптатах от пациентов с метастатическим колоректальным раком в яичник: отчет о болезни». Письма об онкологии. 16 (5): 6431–6436. Дои:10.3892 / ол.2018.9467. ЧВК 6202479. PMID 30405780.
- ^ Класс Д., Ньюман А., Лавджой А.Ф., Чжоу Л., Штер Х, Сюй Т, Хе Дж, Комаки РУ, Ляо З., Мару Д., Ализаде А. (2015). «Анализ циркулирующей опухолевой ДНК у больных карциномой пищевода, получавших химиолучевую терапию». Международный журнал радиационной онкологии * Биология * Физика. 93 (3): S104 – S105. Дои:10.1016 / j.ijrobp.2015.07.251.
- ^ Осмундсон Э, Ньюман А.М., Братман С.В., Класс Д.М., Чжоу Л., Пай Дж., Лонгакр Т.А., Ализаде А.А., Кунг А.С., Дин М. (2014). «Циркулирующая опухолевая ДНК как биомаркер аденокарциномы поджелудочной железы». Международный журнал радиационной онкологии, биологии, физики. 90 (1): S816 – S817. Дои:10.1016 / j.ijrobp.2014.05.2354.
- ^ Дадли Дж.К., Шроерс-Мартин Дж., Лаццарески Д.В., Ши В.Й., Чен С.Б., Исфахани М.С., Триведи Д., Чабон Дж.Дж., Чаудхури А.А., Штер Х., Лю С.Л., Лим Х., Коста Ха, Набет Б.А., Син М.Л., Ляо Дж.С., Ализаде А.А., Дин М. (апрель 2019 г.). «Обнаружение и наблюдение за раком мочевого пузыря с использованием ДНК опухоли мочи». Открытие рака. 9 (4): 500–509. Дои:10.1158 / 2159-8290.CD-18-0825. ЧВК 6467650. PMID 30578357.
- ^ а б Пржибил Дж., Чабон Дж. Дж., Спанс Л., Ганджу К. Н., Веннам С., Ньюман А. М. и др. (Июнь 2018). «Комбинированный подход для обнаружения различных типов изменений в циркулирующей опухолевой ДНК при лейомиосаркоме». Клинические исследования рака. 24 (11): 2688–2699. Дои:10.1158 / 1078-0432.CCR-17-3704. ЧВК 5984700. PMID 29463554.
- ^ Шах А.Т., Азад Т.Д., Чабон Дж.Дж., Бриз М., Танаса Б., Спиллинджер А., Люнг С.Г., Дин М., Ализаде А.А. (01.10.2018). "Abstract B49: Количественное определение циркулирующей опухолевой ДНК у пациентов с транслокационно-положительной саркомой с использованием CAPP-Seq". Стендовые презентации - Предлагаемые тезисы. Американская ассоциация исследований рака: B49. Дои:10.1158 / 1538-7445.PEDCA17-B49.
- ^ Диас Л.А., Барделли А. (февраль 2014 г.). «Жидкая биопсия: генотипирование циркулирующей опухолевой ДНК». Журнал клинической онкологии. 32 (6): 579–86. Дои:10.1200 / JCO.2012.45.2011. ЧВК 4820760. PMID 24449238.
- ^ Хабер Д.А., Велкулеску В.Е. (июнь 2014 г.). «Анализ крови на рак: циркулирующие опухолевые клетки и циркулирующая опухолевая ДНК». Открытие рака. 4 (6): 650–61. Дои:10.1158 / 2159-8290.CD-13-1014. ЧВК 4433544. PMID 24801577.
- ^ а б c d е ж Братман С.В., Ньюман А.М., Ализаде А.А., Дин М. (июнь 2015 г.). «Возможное клиническое применение сверхчувствительного обнаружения циркулирующей опухолевой ДНК с помощью CAPP-Seq». Экспертный обзор молекулярной диагностики. 15 (6): 715–9. Дои:10.1586/14737159.2015.1019476. ЧВК 5052032. PMID 25773944.
- ^ Гримуэйд Д., Вьяс П., Фриман С. (ноябрь 2010 г.). «Оценка минимальной остаточной болезни при остром миелоидном лейкозе». Текущее мнение в области онкологии. 22 (6): 656–63. Дои:10.1097 / CCO.0b013e32833ed831. PMID 20805746. S2CID 205547633.
- ^ Бар-Шалом Р., Ефремов Н., Гуральник Л., Гайтини Д., Френкель А., Кутен А. и др. (Август 2003 г.). «Клиническая эффективность ПЭТ / КТ в оценке рака: дополнительная ценность для диагностической визуализации и ведения пациентов». Журнал ядерной медицины. 44 (8): 1200–9. PMID 12902408.
- ^ Диль Ф., Шмидт К., Чоти М.А., Римлянам К., Гудман С., Ли М. и др. (Сентябрь 2008 г.). «Циркулирующая мутантная ДНК для оценки динамики опухоли». Природа Медицина. 14 (9): 985–90. Дои:10,1038 / нм.1789. ЧВК 2820391. PMID 18670422.
- ^ а б Спина В., Росси Д. (январь 2019 г.). «Жидкая биопсия при тканевых лимфомах». Швейцарский медицинский еженедельник. 149 (23): w14709. Дои:10.4414 / smw.2019.14709. PMID 30673117.