WikiDer > Онкогеномика
Онкогеномика является подполе геномика что характеризует рак-ассоциированный гены. Он фокусируется на геномных, эпигеномных и транскрипционных изменениях при раке.
Рак генетическое заболевание, вызванное накоплением ДНК мутации и эпигенетические изменения, ведущие к безудержной пролиферации клеток и новообразование формирование. Цель онкогеномики - выявление новых онкогены или гены-супрессоры опухолей это может дать новое понимание диагностики рака, прогнозирования клинических исходов рака и новых целей для лечения рака. Успех целевых методов лечения рака, таких как Гливек, Герцептин и Авастин вселили надежду на то, что онкогеномика определит новые цели для лечения рака.[1]
Помимо понимания основных генетических механизмов, которые инициируют или управляют прогрессированием рака, онкогеномика нацелена на индивидуальное лечение рака. Рак развивается из-за случайных мутаций ДНК и эпигенетических изменений. Выявление и выявление мутаций у отдельного пациента может привести к повышению эффективности лечения.
Завершение Проект "Геном человека" способствовал развитию онкогеномики и расширил возможности исследователей в области поиска онкогенов. Технологии секвенирования и методы глобального профилирования метилирования были применены для изучения онкогеномики.
История
Эра геномики началась в 1990-х годах с создания последовательностей ДНК многих организмов. В 21 веке завершение строительства Проект "Геном человека" позволил изучить функциональную геномику и исследовать геномы опухолей. Рак находится в центре внимания.
Эпигеномика началась совсем недавно, примерно в 2000 году.[2][3] Одним из основных источников эпигенетических изменений является измененное метилирование Острова CpG на промоутер область генов (см. Метилирование ДНК при раке). Ряд недавно разработанных методов позволяет оценить статус метилирования ДНК при раке по сравнению с нормальными тканями.[4] Некоторые методы оценивают метилирование CpG, расположенных в разных классах локусов, включая CpG-острова, берега и полки, а также промоторы, тела генов и межгенные области.[5] Рак также находится в центре внимания эпигенетических исследований.
Доступ ко всему секвенирование генома рака важен для исследования рака (или генома рака), потому что:
- Мутации являются непосредственной причиной рака и определяют опухоль фенотип.
- Доступ к образцам раковых и нормальных тканей от одного и того же пациента и тот факт, что большинство мутаций рака представляют собой соматический события, позволяют идентифицировать специфичные для рака мутации.
- Мутации рака накапливаются и иногда связаны со стадией заболевания. Метастаз и лекарственная устойчивость различимы.[6]
Доступ к профилированию метилирования важен для исследований рака, потому что:
- Epi-драйверы, наряду с Mut-driver, могут действовать как непосредственные причины рака.[7]
- Эпимутации рака накапливаются и иногда связаны со стадией заболевания.[8]
Секвенирование всего генома
Первый геном рака был секвенирован в 2008 году.[6] В этом исследовании была проведена последовательность типичных острый миелоидный лейкоз (AML) геном и его нормальный аналог генома получены от одного и того же пациента. Сравнение выявило десять мутировавших генов. Уже считалось, что два фактора способствуют прогрессированию опухоли: внутреннее тандемное дублирование FLT3 рецептор тирозинкиназа ген, который активирует передачу сигналов киназы и связан с плохим прогнозом и вставкой четырех оснований в экзон 12 NPM1 ген (NPMc). Эти мутации обнаруживаются в 25-30% опухолей AML и, как считается, способствуют прогрессированию заболевания, а не вызывают его напрямую.
Остальные 8 были новыми мутациями, и все были изменениями одного основания: четыре были в семьях, которые сильно связаны с патогенезом рака (PTPRT, CDH24, PCLKC и SLC15A1). Остальные четыре ранее не имели связи с патогенезом рака. У них действительно были потенциальные функции в метаболические пути что предложило механизмы, с помощью которых они могут способствовать развитию рака (KNDC1, GPR124, EB12, GRINC1B)
Эти гены участвуют в путях, которые, как известно, вносят вклад в патогенез рака, но до этого исследования большинство из них не могло быть кандидатами для целевой генной терапии. Этот анализ подтвердил подход целого секвенирование генома рака в выявлении соматических мутаций и важности параллельного секвенирования геномов нормальных и опухолевых клеток.[9]
В 2011 году геном исключительного пациента с раком мочевого пузыря, опухоль которого была устранена с помощью препарата. эверолимус был секвенирован, выявив мутации в двух генах, TSC1 и NF2. Мутации нерегулируемые mTOR, белок ингибируется эверолимусом, что позволяет ему воспроизводиться без ограничений. В результате в 2015 году в Национальном институте рака была создана Инициатива исключительных ответчиков. Инициатива позволяет таким исключительным пациентам (которые в течение как минимум шести месяцев положительно реагировали на противораковое лекарство, которое обычно не помогало) секвенировать их геномы для выявления соответствующих мутаций. После идентификации другие пациенты могут быть проверены на наличие этих мутаций, а затем им будет назначено лекарство. В 2016 г. С этой целью в 2015 г. началось общенациональное испытание препарата от рака, в котором приняли участие до двадцати четырехсот центров. Пациентам с соответствующими мутациями подбирают один из более чем сорока препаратов.[10]
В 2014 году Центр молекулярной онкологии запустил тест MSK-IMPACT, инструмент для поиска мутаций в 341 гене, связанном с раком. К 2015 году обследование прошли более пяти тысяч пациентов. Пациенты с соответствующими мутациями имеют право участвовать в клинических испытаниях, обеспечивающих таргетную терапию.[10]
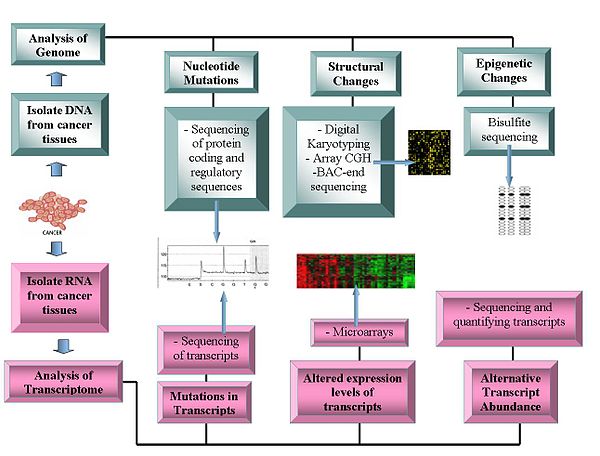
Технологии
Технологии геномики включают:
Секвенирование генома
- Секвенирование ДНК: Пиросеквенированиесеквенсоры на основе предлагают относительно недорогой метод генерации данных последовательности.[1][11][12]
- Сравнительная гибридизация генома: Этот метод измеряет Номер копии ДНК различия между нормальным и раковым геномами. Он использует интенсивность флуоресценции от образцов с флуоресцентной меткой, которые гибридизируются с известными зондами на микрочипе.[13][14]
- Репрезентативный олигонуклеотид микрочип анализ: Обнаруживает изменение количества копий с использованием амплифицированных рестрикционно-переваренных геномных фрагментов, которые гибридизуются с олигонуклеотидами человека, достигая разрешения от 30 до 35 кбит / с.[15]
- Цифровой Кариотипирование: Обнаруживает вариацию количества копий с помощью геномных тегов, полученных с помощью рестрикционный фермент переваривает. Затем эти теги связываются с тегами, объединяются, клонируются, упорядочиваются и сопоставляются обратно с эталонный геном для оценки плотности тегов.[16][17]
- Бактериальная искусственная хромосома (BAC) -концевое секвенирование (профилирование конечной последовательности): Определяет хромосомный точки останова путем создания библиотеки ВАС из генома рака и секвенирования их концов. Клоны ВАС, которые содержат хромосомные аберрации, имеют концевые последовательности, которые не отображаются в аналогичной области эталонного генома, тем самым идентифицируя хромосомную точку разрыва.[18]
Транскриптомы
- Микрочипы: Оценивать стенограмма избыток. Полезно для классификации, прогноза, повышает возможность различных подходов к лечению и помогает идентифицировать мутации в кодирующих областях белков.[19][20] Относительное обилие альтернативных транскриптов стало важной характеристикой исследований рака. Конкретные альтернативные формы транскриптов коррелируют с конкретными типами рака.[21]
- РНК-Seq
Биоинформатика и функциональный анализ онкогенов
Биоинформатика технологии позволяют проводить статистический анализ геномных данных. Функциональные характеристики онкогенов еще предстоит установить. Потенциальные функции включают их трансформационные способности, относящиеся к образованию опухолей и специфические роли на каждой стадии развития рака.
После обнаружения соматический мутации рака в когорте образцов рака, биоинформатический вычислительный анализ может быть проведен для выявления вероятных функциональных и вероятных мутаций драйвера. Для этой идентификации обычно используются три основных подхода: картирование мутаций, оценка влияния мутации на функцию белка или регулирующий элемент и обнаружение признаков положительного отбора в когорте опухолей. Однако подходы не обязательно являются последовательными, существуют важные отношения приоритета между элементами из разных подходов. На каждом этапе используются разные инструменты.[22]
Оперомика
Operomics направлена на интеграцию геномики, транскриптомики и протеомика чтобы понять молекулярные механизмы, лежащие в основе развития рака.[23]
Сравнительная онкогеномика
Сравнительная онкогеномика использует межвидовые сравнения для выявления онкогенов. Это исследование включает изучение геномов, транскриптомов и протеомов рака у модельных организмов, таких как мыши, определение потенциальных онкогенов и обращение к образцам рака человека, чтобы выяснить, важны ли гомологи этих онкогенов в возникновении рака человека.[24] Генетические изменения в моделях мышей аналогичны изменениям, обнаруженным при раке человека. Эти модели создаются с помощью следующих методов: ретровирусный вставка мутагенез или трансплантация трансплантата раковых клеток.
Источник мутаций драйвера рака, мутагенез рака
Мутации обеспечивают исходный материал для естественного отбора в эволюции и могут быть вызваны ошибками репликации ДНК, действием экзогенных мутагенов или эндогенным повреждением ДНК. Механизм репликации и поддержания генома может быть поврежден мутациями или изменен физиологическими условиями и различными уровнями экспрессии при раке (см. Ссылки в [25]).
Как указывает Гао и др.,[26] стабильность и целостность генома человека поддерживаются Ответ на повреждение ДНК (DDR) система. Неправильное повреждение ДНК является основной причиной мутаций, вызывающих канцерогенез.[27][28] Если репарация ДНК недостаточна, повреждения ДНК имеют тенденцию к накоплению. Такое избыточное повреждение ДНК может увеличить мутационный ошибки во время Репликация ДНК из-за подверженности ошибкам транслезионный синтез. Избыточное повреждение ДНК также может увеличиваться эпигенетический изменения из-за ошибок во время ремонта ДНК.[29][30] Такие мутации и эпигенетические изменения могут привести к рак. Гены DDR часто репрессируются при раке человека эпигенетическими механизмами. Такая репрессия может включать метилирование ДНК промоторных областей или репрессию генов DDR с помощью микроРНК. Эпигенетическая репрессия генов DDR происходит чаще, чем мутации генов при многих типах рака (см. Эпигенетика рака). Таким образом, эпигенетическая репрессия часто играет более важную роль, чем мутации, в снижении экспрессии генов DDR. Эта пониженная экспрессия генов DDR, вероятно, является важным фактором канцерогенеза.
Контекст нуклеотидной последовательности влияет на вероятность мутации[31][32][33] и анализ мутационных (изменчивых) мотивов ДНК может иметь важное значение для понимания механизмов мутагенеза при раке. Такие мотивы представляют собой отпечатки взаимодействий между ДНК и мутагенами, между ДНК и ферментами репарации / репликации / модификации. Примерами мотивов являются мотив AID WRCY / RGYW (W = A или T, R = пурин и Y = пиримидин) с мутациями от C до T / G / A,[33] и склонная к ошибкам ДНК pol η приписывала связанные с AID мутации (от A до G / C / G) в мотивах WA / TW.[34]
Другой (независимый) способ анализа наблюдаемых мутационных спектров и контекста последовательности ДНК мутаций в опухолях включает объединение всех мутаций разных типов и контекстов из образцов рака в дискретное распределение. Если доступно несколько образцов рака, их контекстно-зависимые мутации могут быть представлены в виде неотрицательной матрицы. Эта матрица может быть далее разложена на компоненты (мутационные сигнатуры), которые в идеале должны описывать отдельные мутагенные факторы.[35] Было предложено несколько вычислительных методов для решения этой проблемы декомпозиции. Первая реализация метода неотрицательной матричной факторизации (NMF) доступна в Sanger Institute Mutational Signature Framework в виде пакета MATLAB.[36] С другой стороны, если доступны мутации только из одного образца опухоли, пакет DeconstructSigs R[37] и Сервер MutaGene[38] может обеспечить идентификацию вкладов различных мутационных сигнатур для одного образца опухоли. Кроме того, сервер MutaGene предоставляет модели и сигнатуры мутаген-специфичных мутаций, которые могут быть применены для расчета ожидаемой изменчивости сайтов ДНК и белка, чтобы отделить относительный вклад мутагенеза и отбора в канцерогенез.
Синтетическая летальность
Синтетическая летальность возникает, когда комбинация недостатков экспрессии двух или более генов приводит к гибели клеток, тогда как недостаток только одного из этих генов этого не делает. Недостатки могут возникать из-за мутаций, эпигенетических изменений или ингибиторов одного из генов.
Терапевтический потенциал синтетической летальности как эффективной противораковой стратегии постоянно улучшается. В последнее время применение синтетической летальности для таргетной терапии рака возросло благодаря недавней работе ученых, в том числе Рональд А. ДеПиньо и его коллеги, что называется «сопутствующей летальностью». Muller et al. обнаружили, что гены-пассажиры, хромосомно близкие к генам-супрессорам опухолей, в некоторых случаях рака делетируются.[39] Таким образом, идентификация коллатерально удаленных избыточных генов, выполняющих важную клеточную функцию, может быть неиспользованным резервуаром для последующего преследования синтетическая летальность подход. Таким образом, побочная летальность имеет большой потенциал для выявления новых и селективных терапевтических целей в онкологии.[40] В 2012 году Muller et al. определили, что гомозиготная делеция повторяющихся незаменимых гликолитических ENO1 ген в человеке глиобластома (GBM) является следствием близости к делециям локуса опухолевого супрессора 1p36 и может иметь потенциал для синтетического летального подхода к ингибированию GBM.[39] ENO1 является одним из трех гомологичных генов (ENO2, ENO3), который кодирует млекопитающее альфа-енолаза фермент.[41] ENO2, который кодирует энолаза 2, в основном экспрессируется в нервных тканях, что приводит к постулированию, что в ENO1-удаленный GBM, ENO2 может быть идеальной целью в качестве избыточного гомолога ENO1.[42] Мюллер обнаружил, что как генетические, так и фармакологические ENO2 ингибирование в клетках GBM с гомозиготными ENO1 делеция вызывает синтетический летальный исход за счет селективного уничтожения клеток GBM.[39] В 2016 году Мюллер и его коллеги обнаружили антибиотик SF2312 как высокоактивный наномолярный диапазон. энолаза ингибитор, который предпочтительно ингибирует пролиферацию клеток глиомы и гликолитический поток в ENO1-удаленные ячейки.[43] Было показано, что SF2312 более эффективен, чем ингибитор пан-енолазы PhAH, и обладает большей специфичностью в отношении ENO2 торможение над ENO1.[43] Последующая работа той же команды показала, что тот же подход можно применить к панкреатический рак, посредством чего гомозиготно удален SMAD4 приводит к побочной делеции митохондриального яблочного фермента 2 (ME2), окислительной декарбоксилазы, необходимой для редокс гомеостаз.[44] Dey et al. показывают, что геномная делеция ME2 в клетках аденокарциномы протоков поджелудочной железы приводит к высоким уровням эндогенных активных форм кислорода, что согласуется с KRAS-управляемыми панкреатический раки, по существу, заряжает ME2-нулевые клетки для синтетической летальности за счет истощения избыточной NAD (P) + - зависимой изоформы ME3. Было обнаружено, что эффекты истощения ME3 опосредуются ингибированием синтеза нуклеотидов de novo в результате активации AMPK и митохондриального апоптоза, опосредованного ROS.[45][44] Между тем, Oike et al. продемонстрировали обобщаемость концепции, нацелившись на избыточные эссенциальные гены в процессе, отличном от метаболизма, а именно SMARCA4 и SMARCA2 субъединицы ремоделирования хроматина SWI / SNF сложный.[46]
Некоторые онкогены необходимы для выживания всех клеток (не только раковых). Таким образом, препараты, которые уничтожают эти онкогены (и тем самым убивают раковые клетки), также могут повреждать нормальные клетки, вызывая серьезное заболевание. Однако другие гены могут иметь важное значение для раковых клеток, но не для здоровых клеток.
Лечение, основанное на принципе синтетической летальности, продлило выживаемость онкологических больных и обнадеживает будущие успехи в обращении канцерогенеза. Основной тип синтетической летальности воздействует на дефект репарации ДНК, который часто вызывает рак и все еще присутствует в опухолевых клетках. Здесь приведены некоторые примеры.
BRCA1 или BRCA2 экспрессия недостаточна в большинстве случаев рака груди и яичников высокой степени злокачественности, обычно из-за эпигенетического метилирования его промотора или эпигенетической репрессии сверхэкспрессированной микроРНК (см. статьи BRCA1 и BRCA2). BRCA1 и BRCA2 являются важными компонентами основного пути развития гомологичный рекомбинационный ремонт двунитных разрывов. Если один или другой недостаточен, это увеличивает риск рака, особенно рака груди или яичников. Резервный путь репарации ДНК для некоторых повреждений, обычно восстанавливаемых BRCA1 и BRCA2, зависит от PARP1. Таким образом, многие виды рака яичников реагируют на одобренное FDA лечение ингибитором PARP, вызывая синтетическую летальность для раковых клеток, дефицитных по BRCA1 или BRCA2. Это лечение также оценивается для лечения рака груди и многих других видов рака в клинических испытаниях фазы III в 2016 году.[47]
Есть два пути для гомологичный рекомбинационный ремонт двунитных разрывов. Основной путь зависит от BRCA1, PALB2 и BRCA2 в то время как альтернативный путь зависит от RAD52.[48] Доклинические исследования с участием эпигенетически редуцированных или мутировавших BRCA-дефицитных клеток (в культуре или введенных мышам) показывают, что ингибирование RAD52 синтетически летально при дефиците BRCA.[49]
Мутации в генах, используемых в репарации несоответствий ДНК (MMR), вызывают высокую скорость мутаций.[50] В опухолях такие частые последующие мутации часто генерируют «чужие» иммуногенные антигены. В клиническом испытании фазы II с участием 41 пациента оценивали один синтетический летальный подход для опухолей с дефектами MMR или без них.[51] Продукт гена ПД-1 обычно подавляет цитотоксические иммунные ответы. Ингибирование этого гена способствует усилению иммунного ответа. Когда онкологические пациенты с дефектом MMR в их опухолях подвергались воздействию ингибитора PD-1, у 67–78% пациентов наблюдалась иммунная выживаемость без прогрессирования заболевания. Напротив, для пациентов без дефектного MMR добавление ингибитора PD-1 обеспечило только 11% пациентов с иммунной выживаемостью без прогрессирования заболевания. Таким образом, ингибирование PD-1 в первую очередь синтетически летально с дефектами MMR.
ARID1A, модификатор хроматина, необходим для негомологичное соединение концов, основной путь, который восстанавливает двухцепочечные разрывы в ДНК,[52] а также играет роль регулятора транскрипции.[53] Мутации ARID1A - одна из 12 наиболее распространенных канцерогенных мутаций.[54] Мутация или эпигенетически сниженная экспрессия[55] ARID1A обнаружен при 17 типах рака.[56] Доклинические исследования на клетках и на мышах показывают, что синтетическая летальность для дефицита ARID1A возникает либо за счет ингибирования активности метилтрансферазы EZH2, либо[57][58] или с добавлением ингибитора киназы дазатиниба.[59]
Другой подход заключается в индивидуальном отключении каждого гена в геноме и наблюдении за действием на нормальные и раковые клетки.[60][61] Если нокаут несущественного в остальном гена оказывает незначительное влияние на здоровые клетки или совсем не влияет на него, но смертельно опасен для раковых клеток, содержащих мутировавший онкоген, то общесистемное подавление подавленного гена может уничтожить раковые клетки, в то время как здоровые остаются относительно неповрежденными. Методика использовалась для выявления ПАРП-1 ингибиторы для лечения BRCA1/BRCA2-ассоциированные раковые образования.[62][63] В этом случае совместное присутствие ингибирования PARP-1 и связанных с раком мутаций в генах BRCA является летальным только для раковых клеток.
Базы данных для исследований рака
В Проект генома рака это инициатива по выявлению всех соматических мутаций при раке. Проект систематически секвенирует экзоны и фланкирующие сплайсинговые соединения геномов первичных опухолей и линий раковых клеток. Программное обеспечение COSMIC отображает данные, полученные в результате этих экспериментов. По состоянию на февраль 2008 года CGP выявила 4746 генов и 2985 мутаций в 1848 опухолях.
В Проект анатомии генома рака включает информацию об исследованиях раковых геномов, транскриптомов и протеомов.
Progenetix - это онкогеномная справочная база данных, в которой представлены цитогенетические и молекулярно-цитогенетические данные об опухолях.
Oncomine собрал данные из профилей транскриптомов рака.
Интегративная база данных онкогеномики IntOGen и наборы данных Gitools объединяют многомерные онкогеномные данные человека, классифицированные по типу опухоли. Первая версия IntOGen была сосредоточена на роли дерегулируемых экспрессия гена и CNV при раке.[64] Более поздняя версия подчеркнула мутационные гены водителя рака через 28 типов опухолей.[65][66] Все выпуски данных IntOGen доступны в базе данных IntOGen.
В Международный консорциум генома рака это крупнейший проект по сбору данных о геноме рака человека. Данные доступны на веб-сайте ICGC. Пакет BioExpress® Oncology Suite содержит данные об экспрессии генов в образцах первичных, метастатических и доброкачественных опухолей и нормальных образцах, включая соответствующие соседние контроли. В набор входят образцы гематологических злокачественных новообразований для многих известных видов рака.
Конкретные базы данных для модельных животных включают базу данных по генам рака, меченным ретровирусами (RTCGD), в которой собраны исследования по инсерционному мутагенезу ретровирусов и транспозонов в опухолях мышей.
Генные семьи
Мутационный анализ целых семейств генов показал, что гены одного и того же семейства имеют схожие функции, как это предсказывается с помощью аналогичных кодирующих последовательностей и белковые домены. Два таких класса - это киназа семейство, участвующее в добавлении фосфатных групп к белкам и фосфатаза семейство, занимающееся удалением фосфатных групп из белков.[67] Эти семьи были впервые исследованы из-за их очевидной роли в передаче клеточных сигналов о росте или гибели клеток. В частности, более 50% случаев колоректального рака несут мутации в гене киназы или фосфатазы. Фосфатидилинозитолд 3-киназы (PIK3CA) ген кодирует липидкиназы, которые обычно содержат мутации при колоректальном раке, раке молочной железы, желудка, легких и различных других формах рака.[68][69] Медикаментозная терапия может подавлять PIK3CA. Другой пример - BRAF ген, одним из первых вовлеченных в меланомы.[70] BRAF кодирует серин/треонин киназа, которая участвует в RAS-RAF-MAPK сигнальный путь роста. Мутации в BRAF вызывают конститутивное фосфорилирование и активность в 59% меланом. До BRAF генетический механизм развития меланомы был неизвестен, и поэтому прогноз для пациентов был плохим.[71]
Митохондриальная ДНК
Митохондриальная ДНК (мтДНК) мутации связаны с образованием опухолей. Выявлено четыре типа мутаций мтДНК:[72]
Точечные мутации
Точечные мутации наблюдались в кодирующей и некодирующей области мтДНК, содержащейся в раковых клетках. У людей с раком мочевого пузыря, головы / шеи и легких точечные мутации в кодирующей области проявляют признаки сходства друг с другом. Это говорит о том, что когда здоровая клетка трансформируется в опухолевую клетку (неопластическая трансформация), митохондрии кажутся однородными. Обильные точечные мутации, расположенные в некодирующей области, D-петля, раковых митохондрий предполагают, что мутации в этой области могут быть важной характеристикой некоторых видов рака.[72]
Удаления
Этот тип мутации выявляется спорадически из-за его небольшого размера (<1kb). Появление определенных специфических мутаций мтДНК (делеция 264 п.н. и делеция 66 п.н. в гене субъединицы комплекса 1 ND1) при нескольких типах рака является некоторым свидетельством того, что небольшие делеции мтДНК могут появляться в начале туморогенез. Это также предполагает, что количество митохондрий, содержащих эти делеции, увеличивается по мере прогрессирования опухоли. Исключением является относительно большая делеция, которая появляется во многих формах рака (известная как «обычная делеция»), но в нормальных клетках было обнаружено больше крупномасштабных делеций мтДНК по сравнению с опухолевыми клетками. Это может быть связано с кажущимся адаптивным процессом опухолевых клеток по устранению любых митохондрий, содержащих эти крупномасштабные делеции («обычная делеция» составляет> 4kb).[72]
Вставки
Две небольшие вставки мтДНК размером ~ 260 и ~ 520 п.н. могут присутствовать при раке груди, раке желудка, гепатоцеллюлярной карциноме (ГЦК) и раке толстой кишки, а также в нормальных клетках. Корреляции между этими вставками и раком не установлено.[73]
Копирование числовых мутаций
Характеристика мтДНК в режиме реального времени полимеразной цепной реакции Анализы показывают наличие количественного изменения числа копий мтДНК при многих формах рака. Ожидается, что увеличение количества копий произойдет из-за окислительного стресса. С другой стороны, считается, что это снижение вызвано соматическими точечными мутациями в сайте ориджина репликации H-прядь и / или гомополимерный c-участок D310 в области D-петли, мутации в p53 (ген супрессора опухолей) опосредованный путь и / или неэффективная активность фермента из-за ПОЛЬГ мутации. Любое увеличение / уменьшение количества копий остается постоянным в опухолевых клетках. Тот факт, что количество мтДНК в опухолевых клетках постоянно, предполагает, что количество мтДНК контролируется гораздо более сложной системой в опухолевых клетках, а не просто изменяется в результате аномальной пролиферации клеток. Роль содержания мтДНК в раковых заболеваниях человека, по-видимому, варьируется для конкретных типов опухолей или участков.[72]
| Тип рака | Расположение точечных мутаций | Нуклеотидное положение делеций | Увеличение количества копий мтДНК # | Уменьшение копии мтДНК # | |||
|---|---|---|---|---|---|---|---|
| D-петля | мРНК | тРНК | рРНК | ||||
| Мочевой пузырь[74] | Икс | Икс | Икс | 15,642-15,662 | |||
| Грудь[75][76][77][78] | Икс | Икс | Икс | Икс | 8470-13,447 и 8482-13459 | Икс | |
| Голова и шея[75][79][80] | Икс | Икс | Икс | Икс | 8470-13,447 и 8482-13459 | Икс | |
| Устный[81] | Икс | Икс | 8470-13,447 и 8482-13459 | ||||
| Гепатоцеллюлярная карцинома (ГЦК)[82][83] | Икс | Икс | Икс | Икс | 306-556 и 3894-3960 | Икс | |
| Пищевода[84] | Икс | Икс | Икс | 8470-13,447 и 8482-13459 | Икс | ||
| Желудочный [85][86][87] | Икс | Икс | Икс | 298-348 | Икс | ||
| Простата[88][89] | Икс | Икс | 8470-13,447 и 8482-13459 | Икс | |||
57,7% (500/867) содержали соматические точки, а из 1172 обследованных мутаций 37,8% (443/1127) были локализованы в контрольной области D-петли, 13,1% (154/1172) были локализованы в генах тРНК или рРНК и 49,1% (575/1127) были обнаружены в генах мРНК, необходимых для производства комплексов, необходимых для митохондриального дыхания.
Диагностические приложения
Некоторые противоопухолевые препараты нацелены на мтДНК и показали положительные результаты в уничтожении опухолевых клеток. Исследования использовали митохондриальные мутации в качестве биомаркеров для терапии раковых клеток. Нацелить на мутацию внутри митохондриальной ДНК легче, чем на ядерную ДНК, потому что митохондриальный геном намного меньше и его легче проверить на конкретные мутации. Изменения содержания мтДНК, обнаруженные в образцах крови, могут служить маркером скрининга для прогнозирования будущей предрасположенности к раку, а также для отслеживания прогрессирования злокачественной опухоли. Наряду с этими потенциально полезными характеристиками мтДНК, она не находится под контролем клеточного цикла и важна для поддержания АТФ генерация и митохондриальный гомеостаз. Эти характеристики делают нацеливание на мтДНК практической терапевтической стратегией.[72]
Биомаркеры рака
Несколько биомаркеры может быть полезен для определения стадии рака, прогноза и лечения. Они могут варьироваться от однонуклеотидные полиморфизмы (SNP), хромосомные аберрации, изменение числа копий ДНК, микросателлитная нестабильность, промоторная область метилирование, или даже высокий или низкий уровень белка.[90]
Смотрите также
Рекомендации
- ^ а б Страусберг Р.Л., Симпсон А.Дж., Старый Л.Дж., Риггинс Г.Дж. (май 2004 г.). «Онкогеномика и разработка новых методов лечения рака». Природа. 429 (6990): 469–74. Bibcode:2004Натура.429..469S. Дои:10.1038 / природа02627. PMID 15164073.
- ^ Тинг А.Х., МакГарви К.М., Байлин С.Б. (2006). «Эпигеном рака - компоненты и функциональные корреляты». Genes Dev. 20 (23): 3215–31. Дои:10.1101 / gad.1464906. PMID 17158741.
- ^ Джонс П.А., Бейлин С.Б. (2007). «Эпигеномика рака». Клетка. 128 (4): 683–92. Дои:10.1016 / j.cell.2007.01.029. ЧВК 3894624. PMID 17320506.
- ^ Ли Д., Чжан Б., Син Х, Ван Т. (2015). «Объединение MeDIP-seq и MRE-seq для исследования метилирования CpG в масштабе всего генома». Методы. 72: 29–40. Дои:10.1016 / j.ymeth.2014.10.032. ЧВК 4300244. PMID 25448294.
- ^ Вэй Дж, Ли Дж, Данг С., Чжоу Ю, Цзэн К., Лю М. (2016). «Открытие и проверка гиперметилированных маркеров колоректального рака». Дис. Маркеры. 2016: 2192853. Дои:10.1155/2016/2192853. ЧВК 4963574. PMID 27493446.
- ^ а б Страусберг Р.Л., Симпсон А.Дж. (январь 2010 г.). «Полногеномный анализ рака как подход к более глубокому пониманию биологии опухоли». Br. J. Рак. 102 (2): 243–8. Дои:10.1038 / sj.bjc.6605497. ЧВК 2816661. PMID 20029419.
- ^ Фогельштейн Б., Пападопулос Н., Велкулеску В.Э., Чжоу С., Диас Л.А., Кинзлер К.В. (2013). «Пейзажи генома рака». Наука. 339 (6127): 1546–58. Bibcode:2013Научный ... 339.1546V. Дои:10.1126 / наука.1235122. ЧВК 3749880. PMID 23539594.
- ^ Луо Ю., Вонг CJ, Каз AM, Дзецятковски S, Картер К.Т., Моррис С.М., Ван Дж., Уиллис Дж. Э., Макар К. В., Ульрих С. М., Луттербо Дж. Д., Шрубсол MJ, Чжэн В., Марковиц С.Д., Грэди В.М. (2014). «Различия в сигнатурах метилирования ДНК указывают на несколько путей прогрессирования от аденомы к колоректальному раку». Гастроэнтерология. 147 (2): 418–29.e8. Дои:10.1053 / j.gastro.2014.04.039. ЧВК 4107146. PMID 24793120.
- ^ Лей Т.Дж., Мардис Э.Р., Динг Л., Фултон Б., Маклеллан, доктор медицины, Чен К. и др. (Ноябрь 2008 г.). «Секвенирование ДНК цитогенетически нормального генома острого миелоидного лейкоза». Природа. 456 (7218): 66–72. Bibcode:2008Натура 456 ... 66л. Дои:10.1038 / природа07485. ЧВК 2603574. PMID 18987736.
- ^ а б Пейкофф, Кира (2015-10-16). «Что чудесные выздоровления говорят нам о победе над раком». Популярная механика. Получено 2016-04-28.
- ^ Bardelli A .; Велкулеску В.Е. (2005). «Мутационный анализ семейств генов при раке человека». Текущее мнение в области генетики и развития. 15 (1): 5–12. Дои:10.1016 / j.gde.2004.12.009. PMID 15661527.
- ^ Benvenuti S .; Arena S .; Барделли А. (2005). «Идентификация раковых генов путем мутационного профилирования опухолевых геномов». Письма FEBS. 579 (8): 1884–1890. Дои:10.1016 / j.febslet.2005.02.015. PMID 15763568.
- ^ Ши И.М .; Ван Т.Л. (2005). «Применяйте инновационные технологии для изучения генома рака». Текущее мнение в области онкологии. 17 (1): 33–38. Дои:10.1097 / 01.cco.0000147382.97085.e4. PMID 15608510.
- ^ Грешок Дж., Нейлор Т.Л., Марголин А., Дискин С., Кливер С.Х., Futreal PA и др. (Январь 2004 г.). «Сравнительная геномная гибридизация на основе массива с разрешением 1 МБ с использованием набора клонов ВАС, оптимизированного для анализа раковых генов». Genome Res. 14 (1): 179–87. Дои:10.1101 / гр.1847304. ЧВК 314295. PMID 14672980.
- ^ Лучито Р., Хили Дж., Александр Дж., Райнер А., Эспозито Д., Чи М. и др. (Октябрь 2003 г.). «Репрезентативный анализ микроматрицы олигонуклеотидов: метод высокого разрешения для обнаружения вариации числа копий генома». Genome Res. 13 (10): 2291–305. Дои:10.1101 / гр.1349003. ЧВК 403708. PMID 12975311.
- ^ Ху М., Яо Дж, Поляк К. (2006). «Цифровое кариотипирование, специфичное для метилирования». Нат Проток. 1 (3): 1621–36. Дои:10.1038 / nprot.2006.278. PMID 17406428.
- ^ Кёрнер Х., Епанчинцев А., Беркинг С., Шулер-Турнер Б., Speicher MR, Menssen A, Hermeking H (январь 2007 г.). «Цифровое кариотипирование показывает частую инактивацию гена дистрофина / DMD при злокачественной меланоме». Клеточный цикл. 6 (2): 189–98. Дои:10.4161 / cc.6.2.3733. PMID 17314512.
- ^ Волик С., Чжао С., Чин К., Бребнер Дж. Х., Херндон Д. Р., Тао К. и др. (Июнь 2003 г.). «Профилирование конечных последовательностей: анализ аберрантных геномов на основе последовательностей». Proc. Natl. Акад. Sci. СОЕДИНЕННЫЕ ШТАТЫ АМЕРИКИ. 100 (13): 7696–701. Дои:10.1073 / pnas.1232418100. ЧВК 164650. PMID 12788976.
- ^ Van, de Vijver M.J .; и другие. (2002). «Сигнатура экспрессии генов как показатель выживаемости при раке груди». Медицинский журнал Новой Англии. 347 (25): 1999–2009. Дои:10.1056 / NEJMoa021967. PMID 12490681.
- ^ ван 'т Веер Л.Дж., Дай Х., ван де Виджвер М.Дж., Хе Ю.Д., Харт А.А., Бернардс Р., Друг С.Х. (2003). «Профилирование экспрессии предсказывает исход при раке груди». Рак груди Res. 5 (1): 57–8. Дои:10.1186 / bcr562. ЧВК 154139. PMID 12559048.
- ^ Xu Q .; Ли К. (2003). «Открытие новых форм сплайсинга и функциональный анализ рака-специфического альтернативного сплайсинга в человеческих экспрессируемых последовательностях». Исследования нуклеиновых кислот. 31 (19): 5635–5643. Дои:10.1093 / нар / gkg786. ЧВК 206480. PMID 14500827.
- ^ Гонсалес-Перес А., Мустонен В., Рева Б., Ричи Г.Р., Крейкселл П., Карчин Р. и др. (Август 2013). «Вычислительные подходы к идентификации функциональных генетических вариантов в геномах рака». Nat. Методы. 10 (8): 723–9. Дои:10.1038 / nmeth.2562. ЧВК 3919555. PMID 23900255.
- ^ Ханаш С.М. (сентябрь 2000 г.). «Оперомика: молекулярный анализ тканей от ДНК до РНК и белков». Clin. Chem. Лаборатория. Med. 38 (9): 805–13. Дои:10.1515 / CCLM.2000.116. PMID 11097332.
- ^ Пипер Д., Бернс А. (июнь 2006 г.). «Межвидовая онкогеномика в идентификации раковых генов». Клетка. 125 (7): 1230–3. Дои:10.1016 / j.cell.2006.06.018. PMID 16814709.
- ^ Рогозин И.Б., Павлов Ю.И., Гончеаренко А., Де С, Лада А.Г., Поляков Э., Панченко А.Р., Купер Д.Н. (ноябрь 2018). «Мутационные сигнатуры и изменчивые мотивы в геномах рака». Краткий. Биоинформатика. 19 (6): 1085–1101. Дои:10.1093 / bib / bbx049. ЧВК 6454500. PMID 28498882.
- ^ Гао Д, Герман Дж. Г., Го М (2016). «Клиническое значение аберрантных эпигенетических изменений генов восстановления повреждений ДНК при раке человека». Oncotarget. 7 (24): 37331–37346. Дои:10.18632 / oncotarget.7949. ЧВК 5095080. PMID 26967246.
- ^ Кастан МБ (2008). «Реакция на повреждение ДНК: механизмы и роль в человеческих заболеваниях: лекция 2007 г. на присуждении премии имени Г.А. Клоуса». Мол. Рак Res. 6 (4): 517–24. Дои:10.1158 / 1541-7786.MCR-08-0020. PMID 18403632.
- ^ Бернштейн, К; Прасад, АР; Nfonsam, V; Бернштейн, Х. (2013). «Глава 16: Повреждение ДНК, восстановление ДНК и рак». В Чен, Кларк (ред.). Новые направления исследований в области восстановления ДНК. п. 413. ISBN 978-953-51-1114-6.
- ^ О'Хаган Х.М., Мохаммад Х.П., Бейлин С.Б. (2008). «Двухцепочечные разрывы могут инициировать сайленсинг генов и SIRT1-зависимое начало метилирования ДНК в экзогенном промоторном острове CpG». PLoS Genetics. 4 (8): e1000155. Дои:10.1371 / journal.pgen.1000155. ЧВК 2491723. PMID 18704159.
- ^ Куоццо С., Порселлини А., Ангрисано Т. и др. (Июль 2007 г.). «Повреждение ДНК, гомологически направленная репарация и метилирование ДНК». PLoS Genetics. 3 (7): e110. Дои:10.1371 / journal.pgen.0030110. ЧВК 1913100. PMID 17616978.
- ^ Coulondre C, Miller JH, Farabaugh PJ, Gilbert W (август 1978 г.). «Молекулярная основа горячих точек замещения оснований в Escherichia coli». Природа. 274 (5673): 775–80. Bibcode:1978Натура 274..775C. Дои:10.1038 / 274775a0. PMID 355893.
- ^ Купер Д. Н., Юсуфиан Х. (февраль 1988 г.). «Динуклеотид CpG и генетическое заболевание человека». Гм. Genet. 78 (2): 151–5. Дои:10.1007 / bf00278187. PMID 3338800.
- ^ а б Рогозин И.Б., Колчанов Н.А. (ноябрь 1992 г.). «Соматический гипермутагенез в генах иммуноглобулинов. II. Влияние последовательностей соседних оснований на мутагенез». Биохим. Биофиз. Acta. 1171 (1): 11–8. Дои:10.1016 / 0167-4781 (92) 90134-л. ISSN 0006-3002. PMID 1420357.
- ^ Рогозин И.Б., Павлов Ю.И., Бебенек К., Мацуда Т., Кункель Т.А. (июнь 2001 г.). «Горячие точки соматических мутаций коррелируют со спектром ошибок эта ДНК-полимеразы». Nat. Иммунол. 2 (6): 530–6. Дои:10.1038/88732. ISSN 1529-2908. PMID 11376340.
- ^ Ник-Зайнал С., Александров Л.Б., Клин Д.К., Ван Лу П, Гринман С.Д., Рейн К. и др. (Май 2012 г.). «Мутационные процессы, формирующие геномы 21 рака груди». Клетка. 149 (5): 979–93. Дои:10.1016 / j.cell.2012.04.024. ЧВК 3414841. PMID 22608084.
- ^ Александров Л.Б., Ник-Зайнал С, Клин Д.К., Кэмпбелл П.Дж., Страттон М.Р. (январь 2013 г.). «Расшифровка сигнатур мутационных процессов, действующих при раке человека». Сотовый представитель. 3 (1): 246–59. Дои:10.1016 / j.celrep.2012.12.008. ЧВК 3588146. PMID 23318258.
- ^ Розенталь Р., МакГранахан Н., Эрреро Дж., Тейлор Б.С., Суантон С. (февраль 2016 г.). «DeconstructSigs: определение мутационных процессов в единичных опухолях позволяет выявить дефекты репарации ДНК и модели эволюции карциномы». Геном Биол. 17: 31. Дои:10.1186 / s13059-016-0893-4. ЧВК 4762164. PMID 26899170.
- ^ Гончеаренко А., Рейджер С.Л., Ли М., Санг QX, Рогозин И.Б., Панченко А.Р. (июль 2017 г.). «Изучение фоновых мутационных процессов для расшифровки генетической гетерогенности рака». Нуклеиновые кислоты Res. 45 (W1): W514 – W522. Дои:10.1093 / нар / gkx367. ЧВК 5793731. PMID 28472504.
- ^ а б c Muller FL, Colla S, Aquilanti E, Manzo VE, Genovese G, Lee J и др. (Август 2012 г.). «Удаление пассажиров создает терапевтическую уязвимость при раке». Природа. 488 (7411): 337–42. Bibcode:2012Натура.488..337M. Дои:10.1038 / природа11331. ЧВК 3712624. PMID 22895339.
- ^ Мюллер Флорида, Аквиланти Е.А., ДеПиньо РА (ноябрь 2015 г.). «Побочная летальность: новая терапевтическая стратегия в онкологии». Тенденции рака. 1 (3): 161–173. Дои:10.1016 / j.trecan.2015.10.002. ЧВК 4746004. PMID 26870836.
- ^ Пойнер Р. Р., Рид Г. Х. (август 1992 г.). «Структура комплекса двухвалентного катиона с фосфоноацетогидроксаматом в активном центре енолазы». Биохимия. 31 (31): 7166–73. Дои:10.1021 / bi00146a020. PMID 1322695.
- ^ Джозеф Дж., Крус-Санчес Ф.Ф., Каррерас Дж. (Июнь 1996 г.). «Активность энолазы и распределение изоферментов в областях мозга человека и опухолях». J. Neurochem. 66 (6): 2484–90. Дои:10.1046 / j.1471-4159.1996.66062484.x. PMID 8632173.
- ^ а б Леонард П.Г., Сатани Н., Максвелл Д., Лин Ю.Х., Хаммуди Н., Пэн З. и др. (Декабрь 2016 г.). «SF2312 - природный фосфонатный ингибитор енолазы». Nat. Chem. Биол. 12 (12): 1053–1058. Дои:10.1038 / nchembio.2195. ЧВК 5110371. PMID 27723749.
- ^ а б Дей П., Баддур Дж., Мюллер Ф., Ву С.К., Ван Х., Ляо В.Т. и др. (Февраль 2017). «Геномная делеция яблочного фермента 2 вызывает побочную летальность при раке поджелудочной железы». Природа. 542 (7639): 119–123. Bibcode:2017Натура.542..119D. Дои:10.1038 / природа21052. ЧВК 5398413. PMID 28099419.
- ^ Лиу Г.Ю., Депплер Х., Делджорно К.Э., Чжан Л., Лейтжес М., Кроуфорд Х.С., Мерфи М.П., Сторц П. (март 2016 г.). «Мутантный KRas-индуцированный митохондриальный окислительный стресс в ацинарных клетках усиливает сигнализацию EGFR, чтобы управлять образованием предраковых поражений поджелудочной железы». Сотовый представитель. 14 (10): 2325–36. Дои:10.1016 / j.celrep.2016.02.029. ЧВК 4794374. PMID 26947075.
- ^ Оике Т., Огивара Х., Томинага Й., Ито К., Андо О, Цута К. и др. (Сентябрь 2013). «Стратегия, основанная на синтетической летальности, для лечения рака с генетической недостаточностью фактора ремоделирования хроматина BRG1». Рак Res. 73 (17): 5508–18. Дои:10.1158 / 0008-5472.CAN-12-4593. PMID 23872584.
- ^ Мурата С., Чжан С., Финч Н., Чжан К., Кампо Л., Брейер Е.К. (2016). «Предикторы и модуляторы синтетической летальности: новая информация об ингибиторах PARP и персонализированной медицине». Биомед Рес Инт. 2016: 2346585. Дои:10.1155/2016/2346585. ЧВК 5013223. PMID 27642590.
- ^ Лок Б.Х., Карли А.С., Чанг Б., Пауэлл С.Н. (2013). «Инактивация RAD52 является синтетически летальной из-за дефицита в BRCA1 и PALB2 в дополнение к BRCA2 через RAD51-опосредованную гомологичную рекомбинацию». Онкоген. 32 (30): 3552–8. Дои:10.1038 / onc.2012.391. ЧВК 5730454. PMID 22964643.
- ^ Крамер-Моралес К., Неборовска-Скорска М., Шайбнер К., Паджет М., Ирвин Д.А., Сливински Т., Хаас К., Ли Дж., Гэн Х., Рой Д., Слупянек А., Рассул Ф.В., Васик М.А., Чилдерс В., Копленд М., Мюшен М, Цивин К.И., Скорски Т. (2013). «Персонализированная синтетическая летальность, индуцированная нацеливанием на RAD52 при лейкозах, идентифицированных по мутации гена и профилю экспрессии». Кровь. 122 (7): 1293–304. Дои:10.1182 / кровь-2013-05-501072. ЧВК 3744994. PMID 23836560.
- ^ Эшлеман Дж. Р., Ланг Э. З., Бауэрфинд Г. К., Парсонс Р., Фогельштейн Б., Уилсон Дж. К., Вейгл М. Л., Седвик В. Д., Марковиц С. Д. (1995). «Повышенная частота мутаций в локусе hprt сопровождает микросателлитную нестабильность при раке толстой кишки». Онкоген. 10 (1): 33–7. PMID 7824277.
- ^ Ле Д. Т., Урам Дж. Н., Ван Х., Бартлетт Б. Р., Кемберлинг Х., Айринг А. Д. и др. (2015). «Блокада PD-1 в опухолях с недостаточностью восстановления несовпадений». N. Engl. J. Med. 372 (26): 2509–20. Дои:10.1056 / NEJMoa1500596. ЧВК 4481136. PMID 26028255.
- ^ Ватанабэ Р., Уи А., Канно С., Огивара Х., Нагасе Т., Коно Т., Ясуи А. (2014). «Факторы SWI / SNF, необходимые для устойчивости клеток к повреждению ДНК, включают ARID1A и ARID1B и демонстрируют взаимозависимую стабильность белков». Рак Res. 74 (9): 2465–75. Дои:10.1158 / 0008-5472.CAN-13-3608. PMID 24788099.
- ^ Рааб Дж. Р., Резник С., Магнусон Т. (2015). «Общегеномная регуляция транскрипции, опосредованная биохимически различающимися комплексами SWI / SNF». PLoS Genet. 11 (12): e1005748. Дои:10.1371 / journal.pgen.1005748. ЧВК 4699898. PMID 26716708.
- ^ Лоуренс М.С., Стоянов П., Мермел С.Х., Робинсон Дж. Т., Гарравей Л.А., Голуб Т.Р., Мейерсон М., Габриэль С.Б., Ландер Е.С., Гетц Г. (2014). «Обнаружение и анализ насыщения раковых генов по 21 типу опухолей». Природа. 505 (7484): 495–501. Bibcode:2014Натура.505..495л. Дои:10.1038 / природа12912. ЧВК 4048962. PMID 24390350.
- ^ Чжан X, Сун Цюй, Шань М, Ниу М, Лю Т, Ся Би, Лян X, Вэй В, Сунь С, Чжан И, Лю XS, Сон Цюй, Ян И, Ма И, Лю И, Ян Л, Рен Й , Чжан Г, Пан Д (2013). «Гиперметилирование промотора гена ARID1A отвечает за низкую экспрессию его мРНК во многих инвазивных формах рака груди». PLoS ONE. 8 (1): e53931. Bibcode:2013PLoSO ... 853931Z. Дои:10.1371 / journal.pone.0053931. ЧВК 3549982. PMID 23349767.
- ^ Ву Дж. Н., Робертс К. В. (2013). "Мутации ARID1A при раке: еще один эпигенетический супрессор опухоли?". Рак Discov. 3 (1): 35–43. Дои:10.1158 / 2159-8290.CD-12-0361. ЧВК 3546152. PMID 23208470.
- ^ Битлер Б.Г., Эйрд К.М., Гарипов А., Ли Х., Аматанджело М., Косенков А.В., Шульц Д.К., Лю Кью, Ши Ием, Конехо-Гарсия Дж. Р., Спайчер Д. В., Чжан Р. (2015). «Синтетическая летальность за счет воздействия на активность метилтрансферазы EZH2 при раковых опухолях с мутацией ARID1A». Nat. Med. 21 (3): 231–8. Дои:10,1038 / нм.3799. ЧВК 4352133. PMID 25686104.
- ^ Ким К.Х., Ким В., Ховард Т.П., Васкес Ф., Черняк А., Ву Дж. Н., Ван В., Хасвелл Дж. Р., Валенски Л. Д., Хан В. К., Оркин С. С., Робертс К. В. (2015). «Раковые заболевания с мутантным SWI / SNF зависят от каталитической и некаталитической активности EZH2». Nat. Med. 21 (12): 1491–6. Дои:10,1038 / нм.3968. ЧВК 4886303. PMID 26552009.
- ^ Миллер Р. Э., Бро Р., Байрами И., Уильямсон К. Т., Макдейд С., Кэмпбелл Дж. И др. (2016). «Синтетическое летальное нацеливание на ARID1A-мутантные светлоклеточные опухоли яичников с помощью дазатиниба» (PDF). Мол. Рак Ther. 15 (7): 1472–84. Дои:10.1158 / 1535-7163.MCT-15-0554. PMID 27364904.
- ^ Kaelin W.G. (2005). «Концепция синтетической летальности в контексте противоопухолевой терапии». Обзоры природы Рак. 5 (9): 689–698. Дои:10.1038 / nrc1691. PMID 16110319.
- ^ О'Коннор М.Дж .; Martin N.M.B .; Смит Г.К.М. (2007). «Таргетная терапия рака, основанная на подавлении восстановления разрывов цепи ДНК». Онкоген. 26 (56): 7816–7824. Дои:10.1038 / sj.onc.1210879. PMID 18066095.
- ^ Фермер Х., МакКейб Н., Лорд С.Дж., Тутт А.Н., Джонсон Д.А., Ричардсон Т.Б., Сантароса М., Диллон К.Дж., Хиксон И., Найтс С., Мартин Н.М., Джексон С.П., Смит Г.С., Эшворт А. (апрель 2005 г.). «Нацеливание на дефект репарации ДНК в мутантных клетках BRCA как терапевтическая стратегия». Природа. 434 (7035): 917–21. Bibcode:2005Натура.434..917F. Дои:10.1038 / природа03445. PMID 15829967.
- ^ Bryant H.E .; Шульц, Никлас; Thomas, Huw D .; Паркер, Каян М .; Цветок, Дэн; Лопес, Елена; Кайл, Сюзанна; Меут, Марк; Curtin, Nicola J .; Хелледей, Томас; и другие. (2005). «Специфическое уничтожение BRCA2-дефицитных опухолей с помощью ингибиторов поли (АДФ-рибоза) полимеразы». Природа. 434 (7035): 913–917. Bibcode:2005Натура.434..913Б. Дои:10.1038 / природа03443. PMID 15829966.
- ^ Гундем Дж., Перес-Лламас С., Джене-Санз А., Кедзерска А., Ислам А., Деу-Понс Дж., Фурни С. Дж., Лопес-Бигас Н. (февраль 2010 г.). «IntOGen: интеграция и интеллектуальный анализ многомерных онкогеномных данных». Nat. Методы. 7 (2): 92–3. Дои:10.1038 / nmeth0210-92. PMID 20111033.
- ^ Гонсалес-Перес А., Перес-Лламас С., Деу-Понс Дж., Тамбореро Д., Шредер М.П., Джене-Санс А., Сантос А., Лопес-Бигас Н. (ноябрь 2013 г.). «Мутации IntOGen идентифицируют факторы рака среди всех типов опухолей». Nat. Методы. 10 (11): 1081–2. Дои:10.1038 / nmeth.2642. ЧВК 5758042. PMID 24037244.
- ^ Рубио-Перес К., Тамбореро Д., Шредер М.П., Антолин А.А., Деу-Понс Дж., Перес-Лламас К., Местрес Дж., Гонсалес-Перес А., Лопес-Бигас Н. (март 2015 г.). «Назначение противоопухолевых препаратов in silico когортам с 28 типами опухолей открывает возможности для таргетинга». Раковая клетка. 27 (3): 382–96. Дои:10.1016 / j.ccell.2015.02.007. PMID 25759023.
- ^ Блюм-Дженсен П., Хантер Т. (май 2001 г.). «Передача сигналов онкогенной киназы». Природа. 411 (6835): 355–65. Дои:10.1038/35077225. PMID 11357143.
- ^ Bardelli A, et al. (2003). «Мутационный анализ кинома тирозина при колоректальном раке». Наука. 300 (5621): 949. Дои:10.1126 / science.1082596. PMID 12738854.
- ^ Сэмюэлс Й., Ван З., Барделли А., Силлиман Н., Птак Дж., Сабо С., Ян Х., Газдар А., Пауэлл С. М., Риггинс Г. Дж., Уилсон Дж. К., Марковиц С., Кинзлер К. В., Фогельштейн Б., Велкулеску В. Е. (апрель 2004 г.). «Высокая частота мутаций гена PIK3CA при раке человека». Наука. 304 (5670): 554. Дои:10.1126 / science.1096502. PMID 15016963.
- ^ Дэвис Х., Бигнелл Г.Р., Кокс С., Стивенс П., Эдкинс С., Клегг С. и др. (Июнь 2002 г.). «Мутации гена BRAF при раке человека» (PDF). Природа. 417 (6892): 949–54. Bibcode:2002Натура.417..949D. Дои:10.1038 / природа00766. PMID 12068308.
- ^ Danson S .; Лориган П. (2005). «Улучшение результатов при запущенной злокачественной меланоме - обновленная информация о системной терапии». Наркотики. 65 (6): 733–743. Дои:10.2165/00003495-200565060-00002. PMID 15819587.
- ^ а б c d е Ю, Ман (2012). «Соматические мутации митохондриальной ДНК при раке человека». Успехи клинической химии. 57: 99–138. Дои:10.1016 / B978-0-12-394384-2.00004-8. ISBN 9780123943842. PMID 22870588. Цитировать журнал требует
| журнал =(Помогите) - ^ Hung, W.Y .; J.C. Lin; Л. М. Ли; и другие. (2008). «Тандемное дублирование / тройное дублирование коррелирует с вариацией полицитозинового растяжения в области D-петли митохондриальной ДНК человека». Мутагенез. 23 (2): 137–142. Дои:10.1093 / mutage / gen002. PMID 18252697.
- ^ Fliss, M. S .; Usadel, H .; Caballero, O.L .; и другие. (2000). «Быстрое обнаружение мутаций митохондриальной ДНК в опухолях и жидкостях организма». Наука. 287 (5460): 2017–2019. Bibcode:2000Sci ... 287.2017F. Дои:10.1126 / science.287.5460.2017. PMID 10720328.
- ^ а б Дэни, M.A .; S.U. Дани; С.П. Лима; и другие. (2004). «Меньшее количество ΔmtDNA4977, чем обычно, в опухолях различных типов, свидетельствует о том, что раковые клетки практически не подвержены этой мутации». Genet. Мол. Res. 3 (3): 395–409. PMID 15614730.
- ^ Ye, C .; X.O. Шу; W. Wen; и другие. (2008). «Количественный анализ делеции 4977 п.н. митохондриальной ДНК при спорадическом раке груди и доброкачественных заболеваниях груди». Рак молочной железы Res. Рассматривать. 108 (3): 427–434. Дои:10.1007 / s10549-007-9613-9. ЧВК 3836503. PMID 17541740.
- ^ Tseng, L.M .; P.H. Инь; C.W. Chi; и другие. (2006). «Мутации митохондриальной ДНК и истощение митохондриальной ДНК при раке груди». Гены Хромосомы Рак. 45 (7): 629–638. Дои:10.1002 / gcc.20326. PMID 16568452.
- ^ Zhu, W .; В. Цинь; П. Брэдли; А. Вессель; C.L. Пакетт; Э. Р. Заутер (2005). «Мутации митохондриальной ДНК в ткани рака груди и в подобранной аспирационной жидкости из сосков». Канцерогенез. 26 (1): 145–152. Дои:10.1093 / carcin / bgh282. PMID 15375011.
- ^ Zhou S, Kachhap S, Sun W, Wu G, Chuang A, Poeta L, Grumbine L, Mithani SK, Chatterjee A, Koch W., Westra WH, Maitra A, Glazer C, Carducci M, Sidransky D, McFate T, Verma A , Калифано Дж. А (май 2007 г.). «Частота и фенотипические последствия мутаций митохондриальной ДНК при плоскоклеточном раке головы и шеи человека». Proc. Natl. Акад. Sci. СОЕДИНЕННЫЕ ШТАТЫ АМЕРИКИ. 104 (18): 7540–5. Дои:10.1073 / pnas.0610818104. ЧВК 1863503. PMID 17456604.
- ^ Poetsch M, Petersmann A, Lignitz E, Kleist B (март 2004 г.). «Взаимосвязь между нестабильностью митохондриальной ДНК, большими делециями митохондриальной ДНК и нестабильностью ядерных микросателлитов в плоскоклеточных карциномах головы и шеи». Диаг. Мол. Патол. 13 (1): 26–32. Дои:10.1097/00019606-200403000-00005. PMID 15163006.
- ^ Tan, D.J .; Дж. Чанг; W.L. Чен; и другие. (2004). «Соматические мутации митохондриальной ДНК при раке полости рта у жующих бетель-квид». Анна. Акад. Наука. 1011 (1): 310–316. Bibcode:2004НЯСА1011..310Т. Дои:10.1196 / летопись.1293.030. PMID 15126307.
- ^ Lee, H.C .; S.H. Ли; J.C. Lin; C.C. Ву; D.C. Yeh; Y.H. Вэй (2004). «Соматические мутации в D-петле и уменьшение числа копий митохондриальной ДНК при гепатоцеллюлярной карциноме человека». Мутационные исследования. 547 (1–2): 71–78. Дои:10.1016 / j.mrfmmm.2003.12.011. PMID 15013701.
- ^ Инь, P.H .; C.C. Ву; J.C. Lin; C.W. Chi; Y.H. Вэй; H.C. Ли (2010). «Соматические мутации митохондриального генома при гепатоцеллюлярной карциноме». Митохондрия. 10 (2): 174–182. Дои:10.1016 / j.mito.2009.12.147. PMID 20006738.
- ^ Tan, D.J .; Дж. Чанг; Л.Л. Лю; и другие. (2006). «Значение соматических мутаций и изменение содержания митохондриальной ДНК при раке пищевода». BMC Рак. 6: 93. Дои:10.1186/1471-2407-6-93. ЧВК 1459869. PMID 16620376.
- ^ Kassauei K, Habbe N, Mullendore ME, Karikari CA, Maitra A, Feldmann G (2006). «Мутации митохондриальной ДНК при раке поджелудочной железы». Int J Gastrointest Cancer. 37 (2–3): 57–64. Дои:10.1007 / s12029-007-0008-2. PMID 17827523.
- ^ Hung WY, Wu CW, Yin PH, Chang CJ, Li AF, Chi CW, Wei YH, Lee HC (март 2010 г.). «Соматические мутации в митохондриальном геноме и их потенциальная роль в прогрессировании рака желудка человека». Биохим. Биофиз. Acta. 1800 (3): 264–70. Дои:10.1016 / j.bbagen.2009.06.006. PMID 19527772.
- ^ Wu CW, Yin PH, Hung WY, Li AF, Li SH, Chi CW, Wei YH, Lee HC (сентябрь 2005 г.). «Мутации митохондриальной ДНК и истощение митохондриальной ДНК при раке желудка». Гены Хромосомы Рак. 44 (1): 19–28. Дои:10.1002 / gcc.20213. PMID 15892105.
- ^ Yu, J.J .; Т. Ян (2010). «Влияние мутации мтДНК на степень злокачественности опухоли у больных раком простаты». Стареющий мужчина. 13 (3): 159–165. Дои:10.3109/13685530903536668. PMID 20136572.
- ^ Gomez-Zaera, M .; Дж. Абрил; Л. Гонсалес; и другие. (2006). «Идентификация вариантов последовательности ДНК соматических и зародышевых митохондрий у больных раком простаты». Мутационные исследования. 595 (1–2): 42–51. Дои:10.1016 / j.mrfmmm.2005.10.012. PMID 16472830.
- ^ Людвиг Дж. А., Вайнштейн Дж. Н. (ноябрь 2005 г.). «Биомаркеры для определения стадии рака, прогноза и выбора лечения». Nat. Преподобный Рак. 5 (11): 845–56. Дои:10.1038 / nrc1739. PMID 16239904.