WikiDer > Спиральный домен, содержащий 142
В спиральный домен, содержащий 142 (CCDC142) представляет собой ген, который у человека кодирует белок CCDC142. Ген CCDC142 расположен на хромосоме 2 (2p13), охватывает 4339 пар оснований и содержит 9 экзонов. Ген кодирует домен спиральной спирали, содержащий белок 142 (CCDC142), функция которого еще недостаточно изучена.[1][2] Известны две изоформы CCDC142.[1] Белки CCDC142, продуцируемые из этих транскриптов, имеют размер от 743 до 665 аминокислот и содержат сигналы, указывающие на перемещение белка между цитозоль и ядро.[3] Гомологичный Гены CCDC142 обнаружены у многих животных, включая позвоночные и беспозвоночные но нет грибок, растения, протисты, архея, или же бактерии.[1] Хотя функция этого белка не совсем понятна, он содержит домен спиральной спирали и RINT1Мотив _TIP1, расположенный внутри спиральный домен.[3][4]
Locus

CCDC142 обнаружен на - цепи хромосомы 2 (2p13.1) с геномной последовательностью, охватывающей основания от 74 472 832 до 74 483 230.[1] Кодирующая область имеет длину 8292 пары оснований, кодируя две изоформы белка длиной от 743 до 665 аминокислот.[1] На теломерной стороне за CCDC142 следует МОГИ и гены MRPL53. На центромерной стороне за ним следуют C31, LBX2, LBX2-AS1 и PCGF1 гены.[1]
мРНК
В Homo sapiens, ген CCDC142 кодирует две альтернативно сплайсированные изоформы мРНК, называемые изоформой 1 и изоформой 2.[3] Обе эти изоформы имеют 9 экзонов. Изоформа 1 является более длинной из двух и имеет длину 4339 п.н., а изоформа 2 - 2253 п.н.[3] Основное различие между изоформами состоит в том, что изоформа 2 имеет более короткие экзоны 9 и 3 ' UTR.[3] Изоформа 1 - самый длинный вариант гена и белка, которому и посвящена данная статья.[1]
Сохранение
Паралоги
CCDC142 не имеет паралогов в Homo sapiens.
Ортологи
Ниже приведена таблица различных ортологи CCDC142, идентичность белковой последовательности которого сравнивали с Homo sapiens аминокислотная последовательность белка. CCDC142 имеет более 73% аминокислотного сходства в млекопитающие, но менее консервативен в других позвоночные И в беспозвоночные.[5]
| Род и вид | Распространенное имя | Дата отклонения от человеческого происхождения (MYA) | % личность |
| Homo sapiens | Человек | 0 | 100 |
| Пан троглодиты | Шимпанзе | 6.6 | 96 |
| Горилла горилла горилла | Горилла | 8.9 | 98 |
| Jaculus jaculus | Малый египетский тушканчик | 90.9 | 73 |
| Bos mutus | Як | 97.5 | 74 |
| Eptesicus fuscus | Большая коричневая летучая мышь | 97.5 | 74 |
| Питон бивиттатус | Бирманский питон | 320.5 | 36 |
| Gallus gallus | Курица | 320.5 | 35 |
| Haliaeetus leucocephalus | Белоголовый орлан | 320.5 | 33 |
| Анолис каролинский | Каролина аноле (ящерица) | 320.5 | 33 |
| Calidris pugnax | Ерш (птица) | 320.5 | 32 |
| Xenopus tropicalis | Западная когтистая лягушка | 355.7 | 33 |
| Callorhinchus milii | Австралийская акула-призрак | 429.6 | 36 |
| Lepisosteus oculatus | Пятнистый гар | 429.6 | 34 |
| Эсокс Люциус | Северная щука | 429.6 | 33 |
| Данио Рерио | Данио | 429.6 | 33 |
| Lingula anatina | Хвостатая мидия | 847 | 29 |
| Crassostrea gigas | Тихоокеанская устрица | 847 | 29 |
| Осьминог бимакулоидный | Калифорнийский двухточечный осьминог | 847 | 27 |
| Drosophila melanogaster | Плодовая муха | 847 | 23 |
Филогения
CCDC142 имеет близкое родство у млекопитающих, моллюски и амфибии, рептилии и птицы, И в рыбы.[5] Ген CCDC142 появился еще в Drosophila melanogaster, которые отделились от человеческого рода 847 миллионов лет назад. CCDC142 мутировал чаще, чем оба Цитохром с (высококонсервативный белок) и Фибриноген А (быстро мутирующий белок). Это указывает на то, что CCDC142 представляет собой быстро мутирующий ген с увеличивающейся скоростью мутации (то есть эволюции) с течением времени.

Протеин
Первичная структура, варианты и изоформы
Основная изоформа белка CCDC142 имеет длину 743 аминокислоты, а вторая изоформа - 665 аминокислот. Разница в длине полностью обусловлена отсутствием аминокислот на С-конце изоформы 2.[1]
Домены и мотивы
Предсказанный спиральный домен CCDC142 состоит из аминокислот 308-719.[2] А RINT1Мотив _TIP1 также присутствует из аминокислот 490–621. RINT1_TIP1 - это семейство, которое включает RINT-1 (белок, участвующий в радиационно-индуцированном контроле контрольных точек) и TIP-1 (дрожжевой белок, который участвует в Гольджи транспорт).[4] Дополнительные ~ 250 аминокислот, обнаруженные в удаленных белках ортолога CCDC142, не обнаруживаются в Homo sapiens геном ближнего гена CCDC142.
Посттрансляционные модификации
CCDC142, по прогнозам, будет иметь 6 фосфорилирование сайтов, 4 метилирование сайтов, 1 пальмитоилирование сайт, 1 сумоилирование сайт и 1 слабый Сигнал ядерной локализации.[6][7][8][9][10] Эти модификации указывают на то, что CCDC142 локализован в ядро и цитозоль. Аннотации этих сайтов в белке см. В Концептуальном переводе.
Прогнозирование структуры
Вторичная структура CCDC142 содержит только α-спирали как предсказывают программы Quick2D и Phyre2.[11][12] Предполагается, что CCDC142 содержит восемь консервативных α-спирали, шесть из которых расположены в области спиральной спирали белка.[11][12] Предсказанный третичная структура of CCDC142 содержит большой домен coiled-coil из аминокислот 308-719.[2][13]

Выражение
Промоутеры и регулирующие факторы
Промоторная область для CCDC142 была идентифицирована с использованием программы El Dorado в Геноматикс, он охватывает основания 74482896–74483908 в хромосоме 2.[14] Эта область размером 1013 пар оснований охватывает 1071–58 пар оснований перед стартовым кодоном CCDC142.[14] В промоторе есть область, которая связывает большое количество Krueppel-подобные факторы транскрипции и Белки с цинковыми пальцами BED.[14] В этом регионе нет однонуклеотидные полиморфизмы (SNP) расположенные в нем.[15] Многие из факторов транскрипции, которые связываются с промоторной областью CCDC142, имеют функции, связанные с подавлением опухоли, нейрогенез, Повреждение ДНК и фоторецепция.[14] Этот промоторный регион также содержит LTR TATA бокс C-типа млекопитающих который перекрывается с сайтом начала транскрипции гена.[14]
РНК-связывающие белки
Ряд возможных связывающих РНК белков связывается как с 3 ’, так и с 5’ непереведенные регионы (UTRs) мРНК CCDC142. В PABPC1 и RBMX сайты связывания белка встречаются с высокой частотой в 3 ’UTR, с 49 и 21 сайтом соответственно.[16]
Выражение
- Атлас человеческого мозга Аллена Экспрессия CCDC142
Выше Атлас человеческого мозга Аллена данные по экспрессии на CCDC142, красный цвет указывает на более низкую экспрессию, а зеленый - на более высокую.[17] в Homo sapiens головного мозга было обнаружено, что CCDC142 слабо экспрессируется в кора мозжечка, таламус и гипоталамус. CCDC142 также высоко экспрессируется в черная субстанция, мосты, клаустр, и средний мозг.[17] Существует также относительно более высокая экспрессия CCDC142 в рот и вилочковая железа.[18]
- Данные экспрессии NCBI GEO
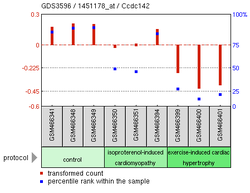
Приведенные выше экспериментальные данные экспрессии показывают много возможных открытий для CCDC142.[19] Сверхэкспрессия SNAI1, а белок цинковых пальцев, коррелирует со снижением экспрессии CCDC142 в Homo sapiens.[20] А Mus musculus выбивной из МЕКК 2/3, который помогает регулировать вспомогательная Т-клетка дифференцировка, также показала пониженную экспрессию CCDC142.[21] Другой Mus musculus эксперимент с упором на кардиомиопатия у мышей показали более низкие уровни CCDC142 у мышей с поврежденным клетки миокарда.[20]
Функция и биохимия
Сочинение
CCDC142 имеет относительно типичное распределение аминокислот по сравнению с другими Homo sapiens белки.[5] Однако в ортологах отмечаются некоторые различия.[5] Лейцин присутствует в больших количествах по сравнению с другими белками (более 15% белка) и аспарагин присутствует в небольших количествах по сравнению с другими белками (менее 0,7% белка).[5]
Домен спиральной спирали и мотив RINT1_TP1 CCDC142 содержат большее количество лейцина по сравнению с остальным белком (более 16,6% области), более высокие количества лейцина. глутамин (более 8,4% от региона) и столь же низкие количества аспарагина (менее 0,7% от региона).[5]
Взаимодействующие белки
Для CCDC142 не обнаружено белковых взаимодействий.
Клиническое значение
Патология и болезни
Увеличение числа копий в локусах CCDC142, включая 25 других генов, показало фенотип задержки развития и значительные онтогенетические или морфологические фенотипы.[22] Один результат с потерей числа копий в локусах CCDC142, включая 29 других генов, показал фенотипы низкого роста, аномальной формы лица, задержки речи и языкового развития, перекрытия пальцев стопы, задержка внутриутробного развития, открытый артериальный протоки задержка развития крупной моторики.[22] Однако влияние CCDC142 могло быть искажено для этих фенотипов, поскольку были также аномалии во многих других геномных секциях.
Мутации
В гене CCDC142 находится ряд SNP. Некоторые из них в промоутер область и 5 ’UTR находятся в якорных последовательностях для факторы транскрипции, и влияют на связывание факторов транскрипции, если они изменены.
В кодирующей последовательности белка есть много SNP, которые изменяют аминокислотный состав CCDC142. Один SNP с высокой степенью распространенности в популяции (1,8%) примечателен своим химическим изменением, с тирозин к сдвигу аспарагина в аминокислоте 548.[15]
Также существует множество SNP, расположенных в большом 3 ’UTR гена, причем многие из них связываются с областями, содержащими структуры петли стебля в мРНК. SNP с распространенностью 7,7% (гуанин к аденозин at bp4285) находится в 3 ’UTR, но не находится в консервативной области петли стебля.[15]
Эти SNP аннотированы в концептуальном переводе, расположенном в разделе «Белки» выше.
Выравнивание нескольких последовательностей
- Выравнивание множественных последовательностей удаленного ортолога CCDC142
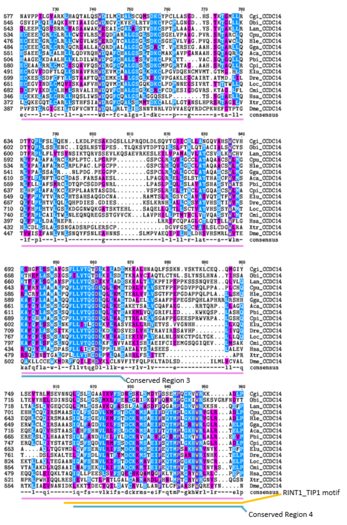
В приведенном выше выравнивании множественной последовательности (созданном с помощью CLUSTALW и программы TEXSHADE в SDSC Biology Workbench) организмы маркируются по первой букве своего рода и первым двум буквам своего вида. Весь белок CCDC142 является высококонсервативным у млекопитающих.[5] Регионы, содержащие Homo sapiens Домен спиральной спирали и область мотива RINT1_TIP1 высоко консервативны в отдаленных гомологах.[5] 12 из 15 аминокислот, которые совпадают у всех организмов в этой области, неполярны.[5] Консервативный регион 1 содержит в основном неполярные аминокислоты.[5] Консервативный регион 2 содержит в основном неполярные и основные аминокислоты. Консервативный регион 3 содержит как полярные, так и неполярные аминокислоты.[5] Консервативный регион 5 содержит в основном неполярные и основные аминокислоты.[5]
Дополнительная информация о факторах транскрипции
Рекомендации
- ^ а б c d е ж грамм час я "CCDC142 домен спиральной спирали, содержащий 142 [Homo sapiens (человек)] - Ген - NCBI". www.ncbi.nlm.nih.gov. Получено 2016-05-01.
- ^ а б c "белок 142, содержащий домен спиральной спирали [Homo sapiens] - белок - NCBI". www.ncbi.nlm.nih.gov. Получено 2016-05-01.
- ^ а б c d е «CCDC142 - белок, содержащий домен спиральной спирали 142 - Homo sapiens (человек) - ген и белок CCDC142». www.uniprot.org. Получено 2016-05-01.
- ^ а б "Результат поиска SSDB Motif: hsa: 84865". www.kegg.jp. Получено 2016-05-01.
- ^ а б c d е ж грамм час я j k л "SDSC Biology Workbench".
- ^ "Сервер NetPhos 2.0". www.cbs.dtu.dk. Получено 2016-05-01.
- ^ "Памятка: Прогноз метилирования белков". www.bioinfo.tsinghua.edu.cn. Архивировано из оригинал на 2016-03-14. Получено 2016-05-01.
- ^ "::: NBA-Palm - Прогнозирование сайта пальмитоилирования, реализованное с помощью наивного байесовского алгоритма :::". www.bioinfo.tsinghua.edu.cn. Архивировано из оригинал на 2016-06-09. Получено 2016-05-01.
- ^ "Программа анализа SUMOplot ™ | Abgent". www.abgent.com. Получено 2016-05-01.
- ^ "NLS_Mapper". nls-mapper.iab.keio.ac.jp. Получено 2016-05-01.
- ^ а б Келли, Лоуренс. "Сервер распознавания складок PHYRE2". www.sbg.bio.ic.ac.uk. Получено 2016-05-01.
- ^ а б Реммерт, Майкл. «Quick2D». toolkit.tuebingen.mpg.de. Получено 2016-05-01.
- ^ а б c «Сервер I-TASSER для предсказания структуры и функции белков». zhanglab.ccmb.med.umich.edu. Получено 2016-05-01.
- ^ а б c d е «Genomatix - Анализ данных NGS и персонализированная медицина». www.genomatix.de. Получено 2016-05-01.
- ^ а б c snpdev. «SNP, связанный с геном (geneID: 84865) через аннотацию Contig». www.ncbi.nlm.nih.gov. Получено 2016-05-01.
- ^ «RBPDB: База данных специфичностей связывания РНК». rbpdb.ccbr.utoronto.ca. Получено 2016-05-01.
- ^ а б "Данные микромассивов :: Атлас мозга Аллена: мозг человека". human.brain-map.org. Получено 2016-05-01.
- ^ "Профиль EST - Hs.430199". www.ncbi.nlm.nih.gov. Получено 2016-05-01.
- ^ гео. «Главная - ГЕО - NCBI». www.ncbi.nlm.nih.gov. Получено 2016-05-01.
- ^ а б "GDS3596 / 1451178_at". www.ncbi.nlm.nih.gov. Получено 2016-05-01.
- ^ "GDS4795 / ILMN_3023885". www.ncbi.nlm.nih.gov. Получено 2016-05-01.
- ^ а б ClinVar. "Ничего не найдено - ClinVar - NCBI". www.ncbi.nlm.nih.gov. Получено 2016-05-05.