WikiDer > TMEM63A
Трансмембранный белок 63A это белок что у людей кодируется TMEM63A ген.[5][6][7] Зрелый человеческий белок составляет примерно 92,1 килодальтон (кДа) с относительно высоким сохранением массы у ортологов.[8] В белке содержится одиннадцать трансмембранные домены и вставляется в мембрану лизосома.[9][10] Анализ BioGPS для TMEM63A у людей показывает, что ген экспрессируется повсеместно, причем самые высокие уровни экспрессии обнаруживаются в Т-клетки и дендритные клетки.[11]
Ген
Обзор
TMEM63A расположен на отрицательной цепи ДНК хромосома 1 в точке 1q42.12, охватывающей пар оснований С 226 033 237 до 226 070 069.[7] Псевдонимы включают KIAA0489 и KIAA0792. Продуктом человеческого гена является 4469 базовая пара мРНК с прогнозируемыми 25 экзоны.[12] Существует 9 прогнозируемых изоформ сплайсинга гена, три из которых кодируют белок. Промоутер анализ проводился с использованием El Dorado[13] сквозь Геноматикс страница программного обеспечения. Предсказанная область промотора охватывает 971 пар оснований, с 226 070 920 до 226 069 950 на отрицательной ветви хромосома 1.
Джин соседство
TMEM63A расположен рядом с EPHX1 ген на положительной смысловой цепи ДНК на хромосома 1, так же хорошо как ЛЕВЫЙ1 ген на отрицательной смысловой цепи.[7] Другие гены в той же области на хромосома 1 включают SRP9 и ЛЕВЫЙ3 на положительной цепи, и МИР6741 и PYCR2 на отрицательной пряди.
Выражение
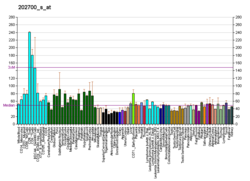
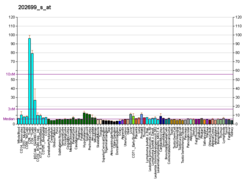
TMEM63 повсеместно экспрессируется в организме человека на разных уровнях, причем наибольшая относительная распространенность встречается у CD 8+ Т-клетки и CD 4+ Т-клетки.[11][14] Умеренные относительные уровни экспрессии также наблюдаются во всем мозге, особенно в затылочная доля, теменная доля, и поджелудочная железа.[14] Анализ TMEM63A экспрессия у мышей с использованием BioGPS выявила больше вариабельных паттернов экспрессии, причем самая высокая экспрессия наблюдалась в желудок и толстая кишка.[11] Использование программы El Dorado от Геноматикс, фактор транскрипции было предсказано, что "TMEM63A" строго регулируется E2F клеточный цикл регуляторы и EGR1, фактор, который считается ген-супрессор опухоли с выражением в мозг.[13] В 3 ’UTR предполагается, что он будет связан регуляторным элементом miR-9 / 9ab.[15]
Протеин
Свойства и характеристики
Зрелая форма человеческого белка TMEM63A имеет 807 аминокислота остатки с изоэлектрическая точка из 6,925.[8] Это довольно хорошо сохраняется в ортологах. А ВЗРЫВ выравнивание показало, что белок содержит три домена: RSN1_TM и два области неизвестной функции (DUF4463 и DUF221).[16] Предполагается, что RSN1_TM будет участвовать в Гольджи везикул транспорт и экзоцитоз. DUF4463 это цитозольный и отдаленно гомологичен РНК-связывающие белки. Этот домен можно использовать для определения ориентации белка в мембрана, с N-конец белка, находящегося в лизосома и C-конец расположен в цитозоль.
Посттрансляционная модификация определено как экспериментально, так и с использованием биоинформатический анализ. Есть два вероятных сайта гликозилирование на белке: N38 и N450.[17] Они были предсказаны с помощью программы NetNGlyc от ExPASy и аминокислотная последовательность TMEM63A, а также предполагаемая ориентация белка в мембране.[18] Есть три вероятных сайта фосфорилирование на белке: S85, S98 и S735, которые были предсказаны с помощью программы NetPhos.[19]
В белке есть три изоформы. Зрелый белок обозначен изоформой CRA. Две другие изоформы - это X1 и X2, которые состоят из 630 аминокислотных остатков и 468 аминокислотных остатков соответственно. Изоформа X1 отсутствует N-конец зрелого белка, в то время как в изоформе 2 отсутствует C-конец.[8]
Взаимодействия
Считается, что, используя текстовую информацию, TMEM63A потенциально может взаимодействовать с шестью другими белками: EEF1D,[20] FAM163B, CPNE9, TMEM90A, STAC2, HEATR3, и WDR67.[21]
Функция
Функция TMEM63A неизвестна, хотя одно исследование показало, что она находится в регионе, который, вероятно, регулируется мир-200а, связанный с эпителиальным гомеостазом.[22] Другой обнаружил, что он находится в локусе количественных признаков, связанных с галоперидол-индуцированный каталепсия.[23]
Эволюционная история
Паралоги
TMEM63A имеет два паралога: TMEM63B, который расположен по адресу 6p21.1, и TMEM63C, который расположен по адресу C14orf171.[24] Соответствие между ними показывает, что TMEM63C более близок к TMEM63B, чем TMEM63A.[8] Выравнивание BLAST показало гомологию TMEM63A и TMEM63B с белками столь же отдаленно родственными, как и растения, тогда как TMEM63C был гомологичен только так же отдаленно, как в дрозофила.[16] Это указывает на то, что TMEM63C, вероятно, расходился с двумя беспозвоночные.
Ортолог пространство
TMEM63A имеет большое пространство ортологов с гомологами, присутствующими в организмах, столь же отдаленно связанных друг с другом, как и у растений.
| Род и виды | Распространенное имя | Учебный класс | Присоединение | Процент идентичности |
|---|---|---|---|---|
| Отолемур гранаттий | Буш ребенок | Млекопитающие | XP_003791028.1 | 91% |
| Vicugna pacos | Альпака | Млекопитающие | XP_006198896.1 | 92% |
| Mus musculus | Мышь | Млекопитающие | NP_659043.1 | 90% |
| Trichechus manatus latirostris | Вест-индийский ламантин | Млекопитающие | XP_004375949.1 | 89% |
| Обыкновенная волчанка | Собака | Млекопитающие | NP_001274088.1 | 89% |
| Myotis davidii | Мышиноухая летучая мышь | Млекопитающие | XP_006761379.1 | 80% |
| Пелодискус китайский | Китайская черепаха софтшелл | Sauropsida | XP_006118107.1 | 71% |
| Аллигатор китайский | Китайский аллигатор | Рептилии | XP_006016630.1 | 70% |
| Ficedula albicollis | Ошейниковая мухоловка | Авес | XP_005043078.1 | 69% |
| Gallus gallus | Красная джунглевая птица | Авес | XP_419384.3 | 68% |
| Xenopus tropicalis | Западная когтистая лягушка | Амфибия | NP_001072343.1 | 65% |
| Ictalurus punctatus | Канальный сом | Актиноптеригии | AHH42519.1 | 54% |
| Culex quinquefasciatus | Комар Южный дом | Насекомое | XP_001861445.1 | 34% |
| Clonorchis sinensis | Китайская печеночная двуустка | Трематода | GAA53916.1 | 23% |
| Oryza sativa | Азиатский рис | Лилиопсида | NP_001065504.1 | 20% |
Рекомендации
- ^ а б c ГРЧ38: Ансамбль выпуск 89: ENSG00000196187 - Ансамбль, Май 2017
- ^ а б c GRCm38: выпуск Ensembl 89: ENSMUSG00000026519 - Ансамбль, Май 2017
- ^ "Справочник человека по PubMed:". Национальный центр биотехнологической информации, Национальная медицинская библиотека США.
- ^ "Ссылка на Mouse PubMed:". Национальный центр биотехнологической информации, Национальная медицинская библиотека США.
- ^ Нагасе Т., Исикава К., Суяма М., Кикуно Р., Миядзима Н., Танака А., Котани Х., Номура Н., Охара О. (апрель 1999 г.). «Прогнозирование кодирующих последовательностей неидентифицированных генов человека. XI. Полные последовательности 100 новых клонов кДНК из мозга, которые кодируют большие белки in vitro». ДНК Res. 5 (5): 277–86. Дои:10.1093 / днарес / 5.5.277. PMID 9872452.
- ^ Секи Н., Охира М., Нагасе Т., Исикава К., Миядзима Н., Накадзима Д., Номура Н., Охара О. (февраль 1998 г.). «Характеристика клонов кДНК в библиотеках кДНК, фракционированных по размеру, из человеческого мозга». ДНК Res. 4 (5): 345–9. Дои:10.1093 / днарес / 4.5.345. PMID 9455484.
- ^ а б c «Ген Entrez: трансмембранный белок 63A TMEM63A».
- ^ а б c d «Анализ TMEM63A». Верстак биологии. Суперкомпьютерный центр Сан-Диего - Калифорнийский университет в Сан-Диего. Получено 8 мая 2014.[постоянная мертвая ссылка]
- ^ Шредер Б.А., Вроклаге С., Хасилик А., Сафтиг П. (19 октября 2010 г.). «Протеом лизосом». Протеомика. 10 (22): 4053–4076. Дои:10.1002 / pmic.201000196. PMID 20957757.
- ^ Schroder BA, Wrocklage C, Pan C, Jager R, Kosters B, Schafer H, Elsasser HP, Mann M, Hasilik A (28 августа 2007 г.). «Интегральные и ассоциированные лизосомальные мембранные белки». Трафик. 8 (12): 1676–1686. Дои:10.1111 / j.1600-0854.2007.00643.x. PMID 17897319.
- ^ а б c «BioGPS: TMEM63A». Получено 12 мая 2014.
- ^ "Ансамбль: TMEM63A". Получено 8 мая 2014.
- ^ а б "Эльдорадо". Геноматикс. Получено 17 апреля 2014.
- ^ а б "GDS596 / 214833_at / TMEM63A". NCBI.
- ^ "TargetScanHuman 6.2". Институт биомедицинских исследований Уайтхеда. Получено 23 апреля 2014.
- ^ а б Марчлер-Бауэр А. и др. (2011). «CDD: база данных консервативных доменов для функциональной аннотации белков». Нуклеиновые кислоты Res. 39 (D): 225–229. Дои:10.1093 / nar / gkq1189. ЧВК 3013737. PMID 21109532.
- ^ "O94886 (TM63A_HUMAN)". UniProtKB. Получено 5 мая 2014.
- ^ Гупта Р., Юнг Э, Брунак С. (2004). «Прогнозирование сайтов N-гликозилирования в человеческих белках». Цитировать журнал требует
| журнал =(помощь) - ^ Блорн Н., Гаммельтофт С., Брунак С. (1999). «Прогнозирование на основе последовательности и структуры сайтов фосфорилирования эукариотических белков». Журнал молекулярной биологии. 294 (5): 1351–1362. Дои:10.1006 / jmbi.1999.3310. PMID 10600390.
- ^ «Генные карты». Институт науки Вейцмана. Получено 16 мая 2014.
- ^ «Строковая база данных». Получено 16 мая 2014.
- ^ Боннет Э, Татари М., Джоши А. и др. (2010). «Вывод сети модулей из набора данных об экспрессии ракового гена позволяет идентифицировать модули, регулируемые микроРНК». PLoS ONE. 5 (4): e10162. Дои:10.1371 / journal.pone.0010162. ЧВК 2854686. PMID 20418949.
- ^ Хофстеттер Дж. Р., Хитземанн Р. Дж., Белкнап Дж. К., Вальтер Н. А., МакВини С. К., Майеда А. Р. (2008). «Характеристика локуса количественного признака галоперидол-индуцированной каталепсии на дистальной хромосоме 1 мыши». Гены, мозг и поведение. 7 (2): 214–223. Дои:10.1111 / j.1601-183x.2007.00340.x. PMID 17696997.
- ^ Альтшул С.Ф., Гиш В., Миллер В., Майерс Е. В., Липман Д. Д. (октябрь 1990 г.). «Базовый инструмент поиска локального выравнивания». J. Mol. Биол. 215 (3): 403–10. Дои:10.1016 / S0022-2836 (05) 80360-2. PMID 2231712.
дальнейшее чтение
- Грегори С.Г., Барлоу К.Ф., Маклей К.Э. и др. (2006). «Последовательность ДНК и биологическая аннотация хромосомы 1 человека». Природа. 441 (7091): 315–21. Дои:10.1038 / природа04727. PMID 16710414.
- Герхард Д.С., Вагнер Л., Фейнгольд Е.А. и др. (2004). «Статус, качество и расширение проекта NIH полноразмерной кДНК: Коллекция генов млекопитающих (MGC)». Genome Res. 14 (10B): 2121–7. Дои:10.1101 / гр.2596504. ЧВК 528928. PMID 15489334.
- Ота Т., Сузуки Ю., Нисикава Т. и др. (2004). «Полное секвенирование и характеристика 21 243 полноразмерных кДНК человека». Nat. Genet. 36 (1): 40–5. Дои:10,1038 / ng1285. PMID 14702039.
- Strausberg RL, Feingold EA, Grouse LH, et al. (2003). «Создание и первоначальный анализ более 15 000 полноразмерных последовательностей кДНК человека и мыши». Proc. Natl. Акад. Sci. СОЕДИНЕННЫЕ ШТАТЫ АМЕРИКИ. 99 (26): 16899–903. Дои:10.1073 / pnas.242603899. ЧВК 139241. PMID 12477932.
- Судзуки Ю., Ёситомо-Накагава К., Маруяма К. и др. (1997). «Создание и характеристика полноразмерной библиотеки кДНК, обогащенной по 5'-концу». Ген. 200 (1–2): 149–56. Дои:10.1016 / S0378-1119 (97) 00411-3. PMID 9373149.
- Маруяма К., Сугано С. (1994). «Олиго-кэппинг: простой метод замены кэп-структуры эукариотических мРНК олигорибонуклеотидами». Ген. 138 (1–2): 171–4. Дои:10.1016/0378-1119(94)90802-8. PMID 8125298.