WikiDer > PETase - Википедия
| ПЭТаза | |||||||||
|---|---|---|---|---|---|---|---|---|---|
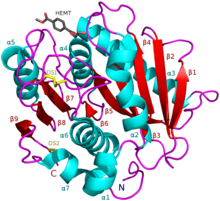 I. sakaiensis ПЭТаза (A0A0K8P6T7) в комплексе с НЕМТ, аналогом ПЭТ (PDB: 5XH3). | |||||||||
| Идентификаторы | |||||||||
| Номер ЕС | 3.1.1.101 | ||||||||
| Альт. имена | ПЭТ гидролаза, поли (этилентерефталат) гидролаза | ||||||||
| Базы данных | |||||||||
| IntEnz | Просмотр IntEnz | ||||||||
| БРЕНДА | BRENDA запись | ||||||||
| ExPASy | Просмотр NiceZyme | ||||||||
| КЕГГ | Запись в KEGG | ||||||||
| MetaCyc | метаболический путь | ||||||||
| ПРИАМ | профиль | ||||||||
| PDB структуры | RCSB PDB PDBe PDBsum | ||||||||
| |||||||||
ПЭТ являются эстераза класс ферменты который катализировать то гидролиз из полиэтилентерефталат (ПЭТ) пластик в мономерный моно-2-гидроксиэтил терефталат (MHET). Идеализированная химическая реакция (где n - количество мономеры в полимер цепь):[1]
- (этилентерефталат)п + H2O → (этилентерефталат)п-1 + MHET
Следы количества ПЭТ распадаются на бис (2-гидроксиэтил) терефталат (BHET). ПЭТ-чехлы также могут разрушать ПЭФ-пластик (полиэтилен-2,5-фурандикарбоксилат), который является заменой ПЭТ на основе биопрепаратов. ПЭТазы не могут катализировать гидролиз алифатический полиэфиры подобно полибутиленсукцинат или же полимолочная кислота.[2]
Неферментативное естественное разложение ПЭТ займет сотни лет, но ПЭТазы могут разложить ПЭТ за считанные дни.[3]
История
Первая ПЭТаза была обнаружена в 2016 году из Ideonella sakaiensis штамм 201-F6 бактерии обнаружены в образцах осадка, собранных недалеко от японского завода по переработке ПЭТ-бутылок.[1][4] Другие виды деградации ПЭТ гидролазы были известны до этого открытия.[2] К ним относятся такие гидролазы, как липазы, эстеразы и кутиназы.[5] Открытия полиэстер деградирующие ферменты датируются как минимум 1975 г. (α-химотрипсин)[6] и 1977 (липаза) Например.[7]
ПЭТ-пластик получил широкое распространение в 1970-х годах, и было высказано предположение, что ПЭТазы в бактериях появились только недавно.[2] В прошлом ПЭТаза могла обладать ферментативной активностью, связанной с разрушением воскового покрытия растений.[8]
Структура
По состоянию на апрель 2019 года известно 17 трехмерных кристаллических структур ПЭТаз: 6QGC, 6ILX, 6ILW, 5YFE, 6EQD, 6EQE, 6EQF, 6EQG, 6EQH, 6ANE, 5XJH, 5YNS, 5XFY, 5XFZ, 5XG0, 5XH2 и 5XH3.
ПЭТаза обладает общими качествами как с липазами, так и с кутиназами в том, что она обладает α / β-гидролаза складывать; хотя расщелина активного центра, наблюдаемая в ПЭТазе, более открыта, чем в кутиназах.[2] В Ideonella sakaiensis ПЭТаза похожа на диенелактонгидролазу, согласно Pfam. Согласно ESTHER, он относится к семейству полиэстераза-липаза-кутиназа.
Существует приблизительно 69 ферментов, подобных ПЭТазе, содержащих множество различных организмов, и существует две классификации этих ферментов, включая тип I и тип II. Предполагается, что 57 ферментов относятся к категории I типа, тогда как остальные относятся к группе II типа, включая фермент ПЭТаза, обнаруженный в Ideonella sakaiensis. Во всех 69 ферментах, подобных ПЭТазе, существуют одни и те же три остатка в активном центре, что позволяет предположить, что каталитический механизм одинаков для всех форм ферментов, подобных ПЭТазе.[9]
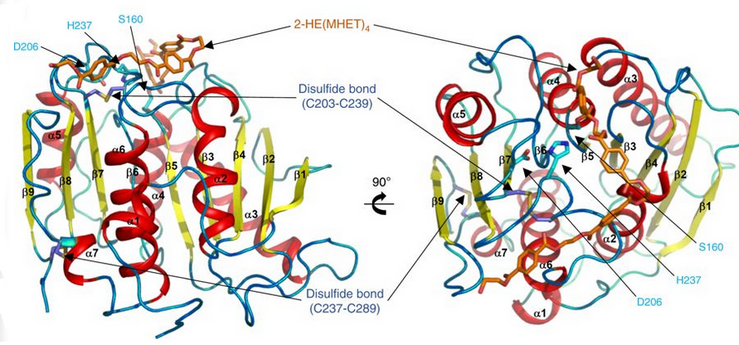
Поверхность PETase double мутант (р103грамм и S131А) с HEMT (1- (2-гидроксиэтил) 4-метилтерефталат), связанным с его активный сайт. HEMT является аналогом MHET и имеет дополнительный метанол этерифицированный к нему. PDBID: 5XH3.
Ленточная диаграмма ПЭТазы с тремя остатками Ser160, Asp206 и His237. Каталитическая триада представлена палочками голубого цвета. Активный сайт показан оранжевым, что означает стимуляцию 2-HE (MHET).4 молекула.[9]
Мутации
В 2018 году ученые из Портсмутского университета в сотрудничестве с Национальной лабораторией возобновляемых источников энергии Министерства энергетики США разработали мутант этой ПЭТазы, который разлагает ПЭТ быстрее, чем в его естественном состоянии. В этом исследовании также было показано, что ПЭТазы могут разлагать полиэтилен 2,5-фурандикарбоксилат (PEF).[2][10]
Биологический путь

В I. sakaiensis, результирующий MHET далее разбивается под действием MHETase фермент терефталевая кислота и этиленгликоль.[1] Лабораторные эксперименты показали, что химерные белки, которые искусственно связывают MHETase и PETase, превосходят аналогичные смеси свободных ферментов.[12]
Смотрите также
| Искать Petase в Викисловаре, бесплатном словаре. |
- Организмы, разрушающие пластик
- Galleria mellonella, гусеница, которая может переваривать полиэтилен.
- Aspergillus tubingensis, гриб, который может переваривать полиуретан.
- Pestalotiopsis microspora, эндофитный гриб, способный расщеплять полиуретан.
- кутиназа, фермент эстераза аналогичной геометрической формы
Рекомендации
- ^ а б c Йошида С., Хирага К., Такехана Т., Танигучи И., Ямаджи Х., Маэда Ю. и др. (Март 2016 г.). «Бактерия, которая разлагает и ассимилирует поли (этилентерефталат)». Наука. 351 (6278): 1196–9. Дои:10.1126 / science.aad6359. PMID 26965627. Сложить резюме (PDF) (2016-03-30).
- ^ а б c d е Остин HP, Аллен MD, Донохо Б.С., Роррер Н.А., Кернс Флорида, Силвейра Р.Л. и др. (Май 2018). «Характеристика и разработка разлагающей пластики ароматической полиэстеразы». Труды Национальной академии наук Соединенных Штатов Америки. 115 (19): E4350 – E4357. Дои:10.1073 / pnas.1718804115. ЧВК 5948967. PMID 29666242.
- ^ Докрил, Питер. «Ученые случайно создали мутантный фермент, который поедает пластиковые отходы». ScienceAlert. Получено 2018-11-27.
- ^ Танасупават С., Такехана Т., Йошида С., Хирага К., Ода К. (август 2016 г.). «Ideonella sakaiensis sp. Nov., Выделенная из микробного консорциума, разлагающего поли (этилентерефталат)». Международный журнал систематической и эволюционной микробиологии. 66 (8): 2813–8. Дои:10.1099 / ijsem.0.001058. PMID 27045688.
- ^ Хан X, Лю В., Хуанг Дж. В., Ма Дж., Чжэн Ю., Ко Т.П. и др. (Декабрь 2017 г.). «Структурное понимание каталитического механизма ПЭТ-гидролазы». Nature Communications. 8 (1): 2106. Дои:10.1038 / s41467-017-02255-z. ЧВК 5727383. PMID 29235460.
- ^ Табуши I, Ямада Х, Мацузаки Х, Фурукава Дж (август 1975 г.). «Полиэстер легко гидролизуется химотрипсином». Журнал науки о полимерах: издание полимерных писем. 13 (8): 447–450. Дои:10.1002 / pol.1975.130130801.
- ^ Tokiwa Y, Suzuki T (ноябрь 1977 г.). «Гидролиз полиэфиров липазами». Природа. 270 (5632): 76–8. Дои:10.1038 / 270076a0. PMID 927523.
- ^ «Лаборатория« Несчастный случай »превратилась в мутантный фермент, поедающий пластик». Живая наука. Получено 2018-11-27.
- ^ а б Джу С., Чо АйДжей, Со Х., Сон Х.Ф., Сагонг Х.Й., Шин Т.Дж. и др. (Январь 2018). «Структурное понимание молекулярного механизма деградации полиэтилентерефталата». Nature Communications. 9 (1): 382. Дои:10.1038 / s41467-018-02881-1. ЧВК 5785972. PMID 29374183.
- ^ Кэррингтон, Дамиан. «Новый суперфермент поедает пластиковые бутылки в шесть раз быстрее». Хранитель.
- ^ Эллисон Чан (2016). «Будущее бактерий, очищающих наши пластиковые отходы» (PDF).
- ^ Knott BC, Erickson E, Allen MD, Gado JE, Graham R, Kearns FL, et al. (Октябрь 2020 г.). «Характеристика и разработка двухферментной системы для деполимеризации пластмасс». Proc Natl Acad Sci U S A. 117 (41): 25476–25485. Дои:10.1073 / pnas.2006753117. PMID 32989159.